相关性分析与PPI网络的构建1、相关性分析我们做完了差异分析之后,我们把上调最显著的20个基因和下调最显著的20个基因进行相关性分析。当然我们在做这个分析的时候,我们是上调和下调都超过了20个的时候。我们才能把上调最显著的和下调最显著的20个挑出来做分析。我们做分析的时候,有时候我们做的上调有35个基因,下调只有15个基因。那我们做这个图的话,那我们就可以在上调里面把最显著的20个基因挑出来。但是下调的时候,他只有15个,那我们就把这15个基因挑出来做这样的相关性分析的图形。下面我们看一下这个图,这个图的话,它的横纵坐标都是基因,并且他们是完全一致的,顺序名字都是一致的。然后在对角线上两个基因,自己和自己比较,所以他们的相关性是最强的,这里的红色的点就代表这两个基因之间是正相关的关系,然后绿色的点就代表这两个基因是负相关的关系。我们主要可以找出我们的目标基因,找到了我们的目标基因之后,我们就可以看一下我们的目标基因跟哪些基因是正相关的,跟哪些基因是负相关的,并且可以通过颜色的深浅判断相关性的强弱,颜色越深相关性越强。  下面我们来看一下临床相关性分析需要的脚本和输入文件,输入文件的话我们需要得到差异分析相关性文件,这个在我们之前的文章中已经得到了就是corInput.txt,还有就是生信自学网提供的脚本文件。  用R运行脚本文件,我们就可以得到我们相关性分析的结果图形。 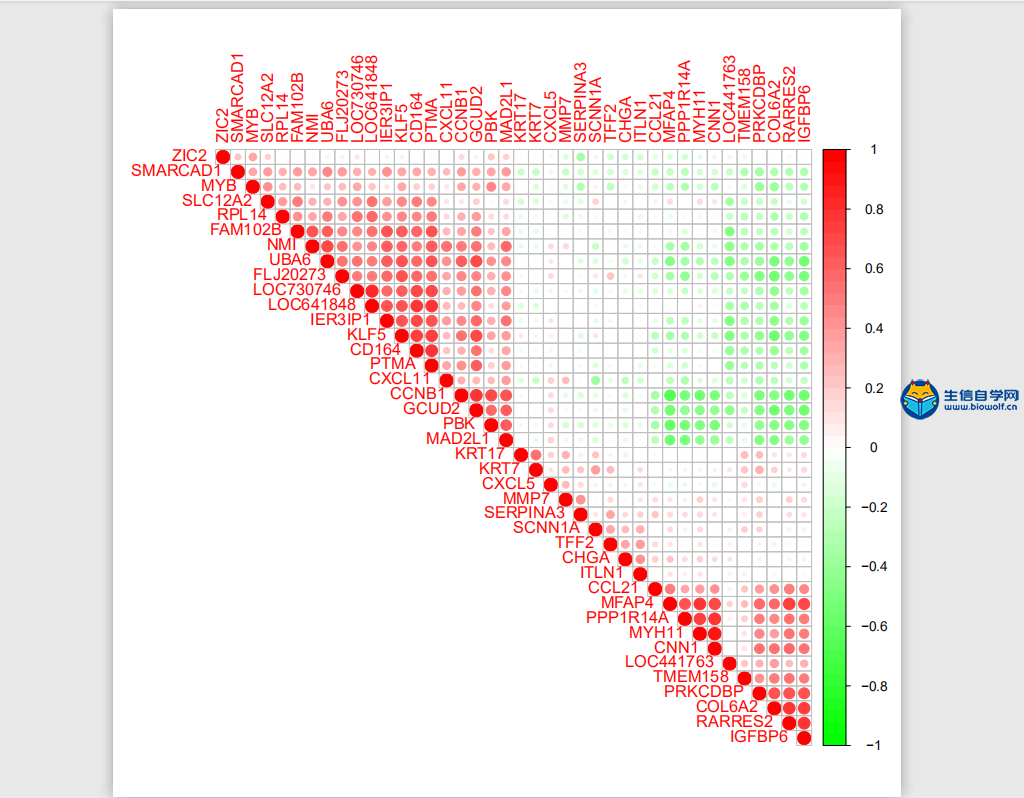 2、PPI网络的构建 我们得到了差异基因之后,我们就可以用这些差异基因。去构建蛋白互作网络,我们就可以得到这样一个图形。这个图形的圆圈就代表我们的蛋白,如果两个蛋白之间有连线的话,就说明这两个蛋白之间具有相互作用的关系。 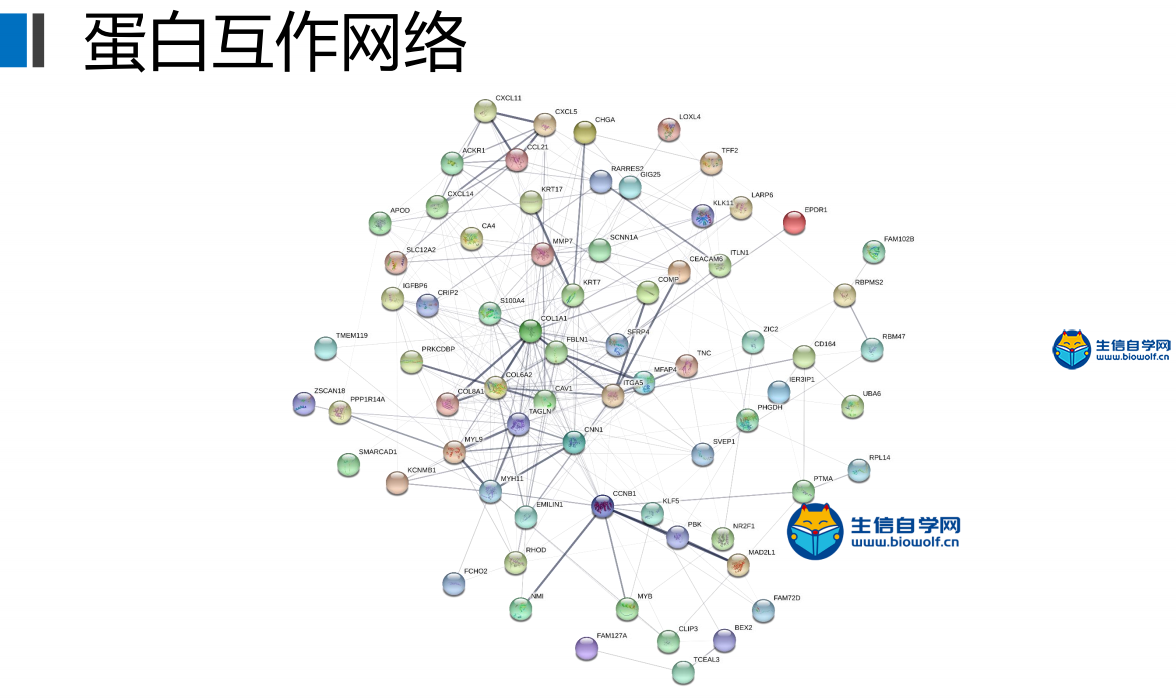 接下来构建蛋白互作网络,我们需要的输入文件是差异分析得到的差异基因文件,这在我们做差异分析的时候已经得到了。接着打开浏览器,搜索STRING PPI,进入网页http://string-db.org/cgi/input.pl?sessionId=bQd8eIWGtELS&input_page_show_search=on 将差异基因文件里的基因名复制粘贴到如下图所示的位置,选择好要研究的对象,点击开始,等待网页加载完成,在最后面点击继续按钮,这样我们就可以得到我们的蛋白互作网络图形。  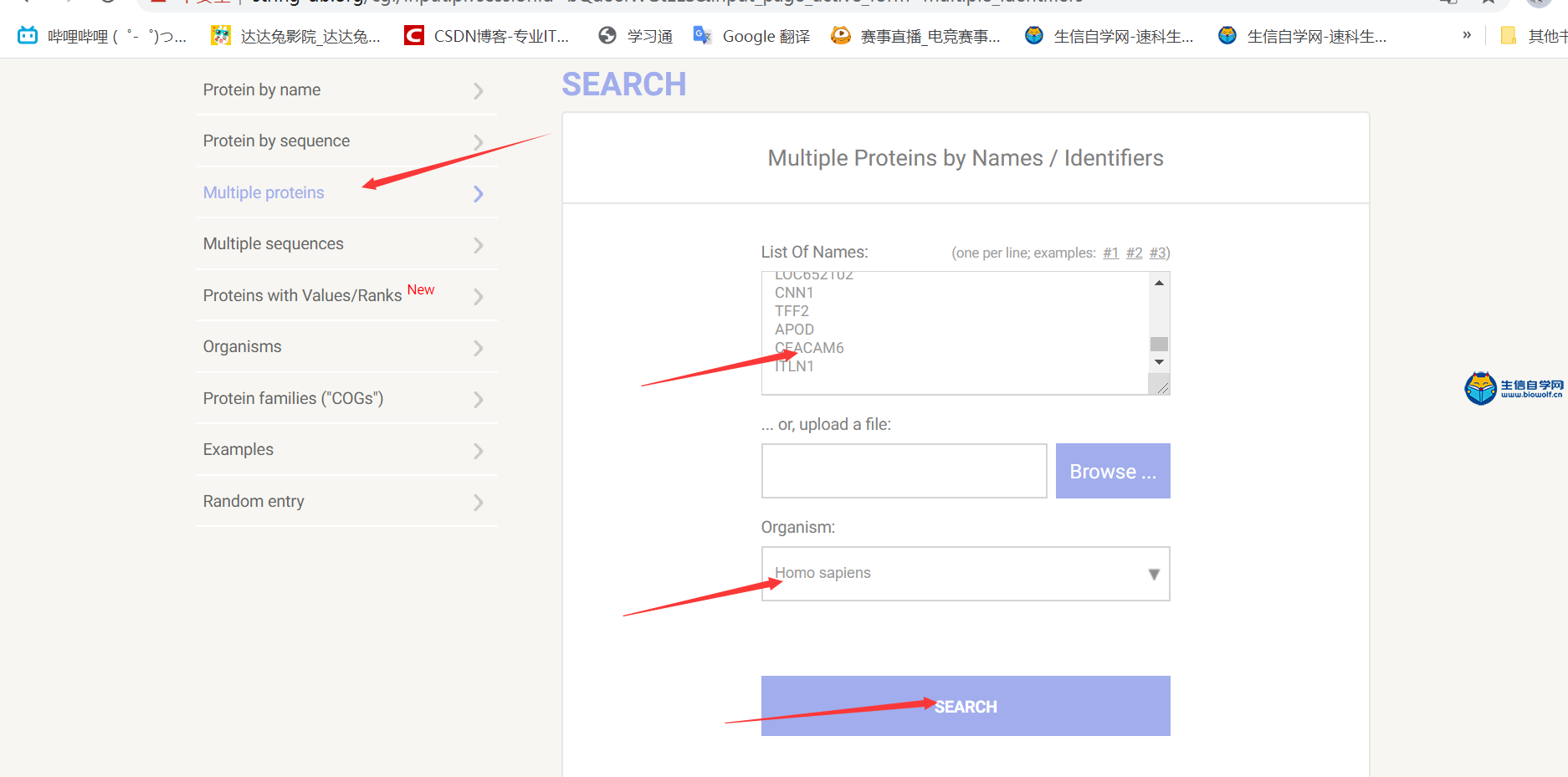 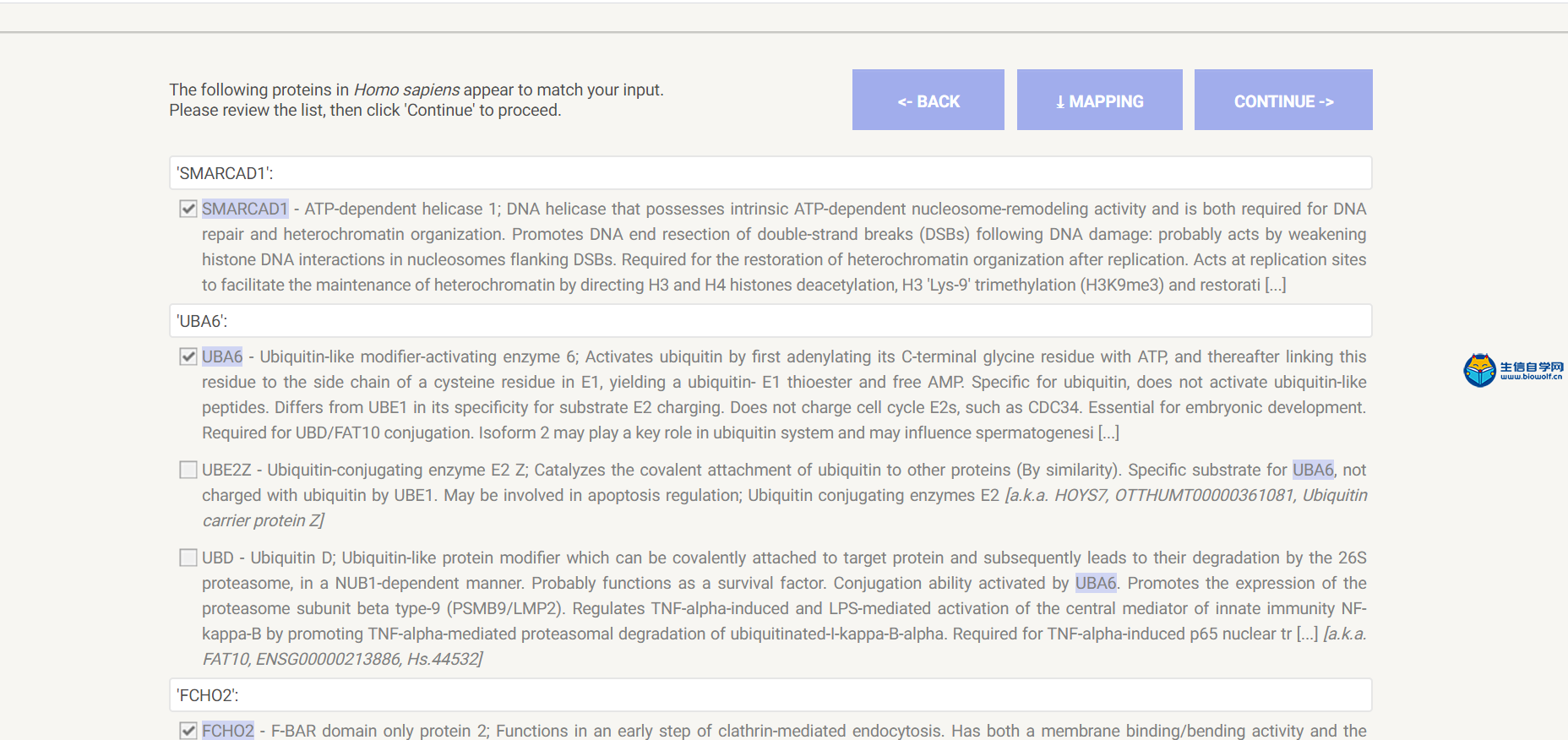 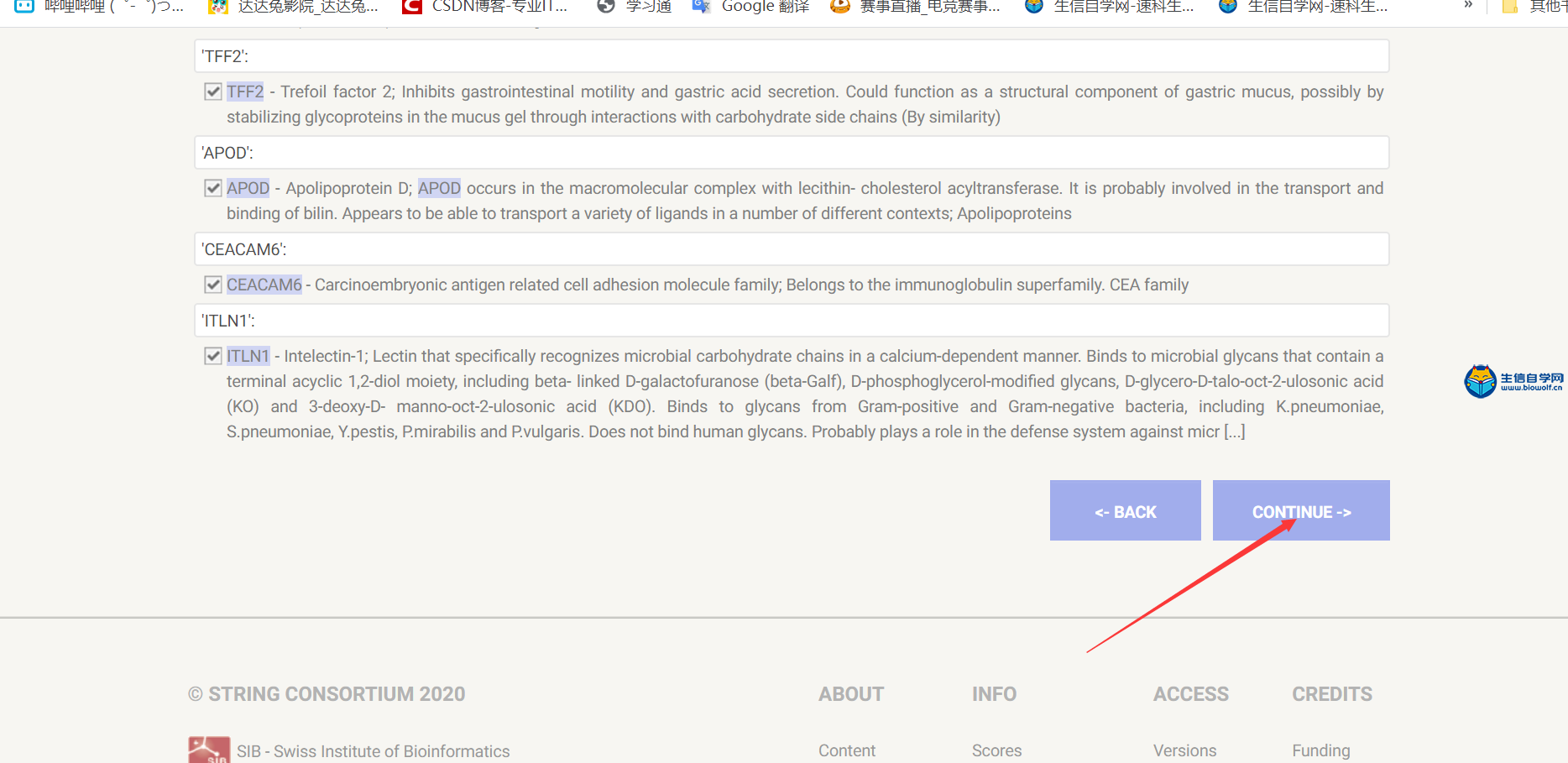 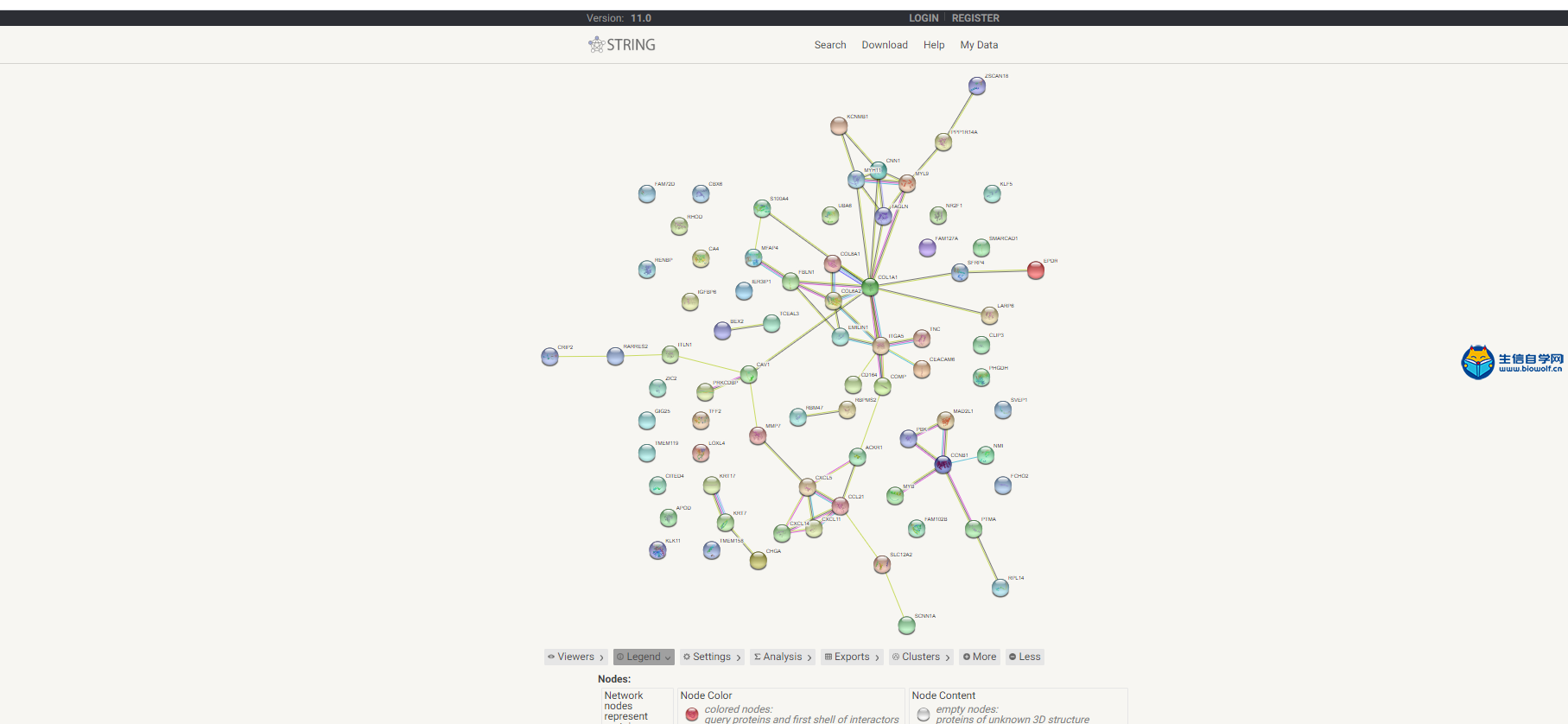 精品课程推荐: 《GEO基础课程》 《GEO数据库单基因挖掘文章套路》 《GEO数据库单细胞测序》  
责任编辑:伏泽 作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载! |





