单基因的差异分析与单基因的配对差异分析差异分析 上一篇文章中,我们已经找到了预后相关的核心基因,之后,我们可以用我们的得到的预后相关的核心基因进行后续的单基因的套路分析,我们首先可以做单基因的差异分析。通过单基因的差异分析我们可以得到如下这样一个图形,在这个图形里面,它的横坐标代表的是样品的类型,左边是正常的样品,右边是肿瘤的样品。然后纵坐标就是我们选取的目标基因的表达量,然后我们可以比较这个基因在正常组和肿瘤组是否具有差异,我们就可以看这个Pvalue。如果P小于0.05,就说明这个基因在正常组和肿瘤组是具有差异的,同时通过这个图我们也可以看出我们这个基因他在正常组里面是低表达的,在肿瘤组里面是高表达的,就说明这个基因在肿瘤组里面是上调的,就可以很直观的看出这个基因的一个上下调的关系。 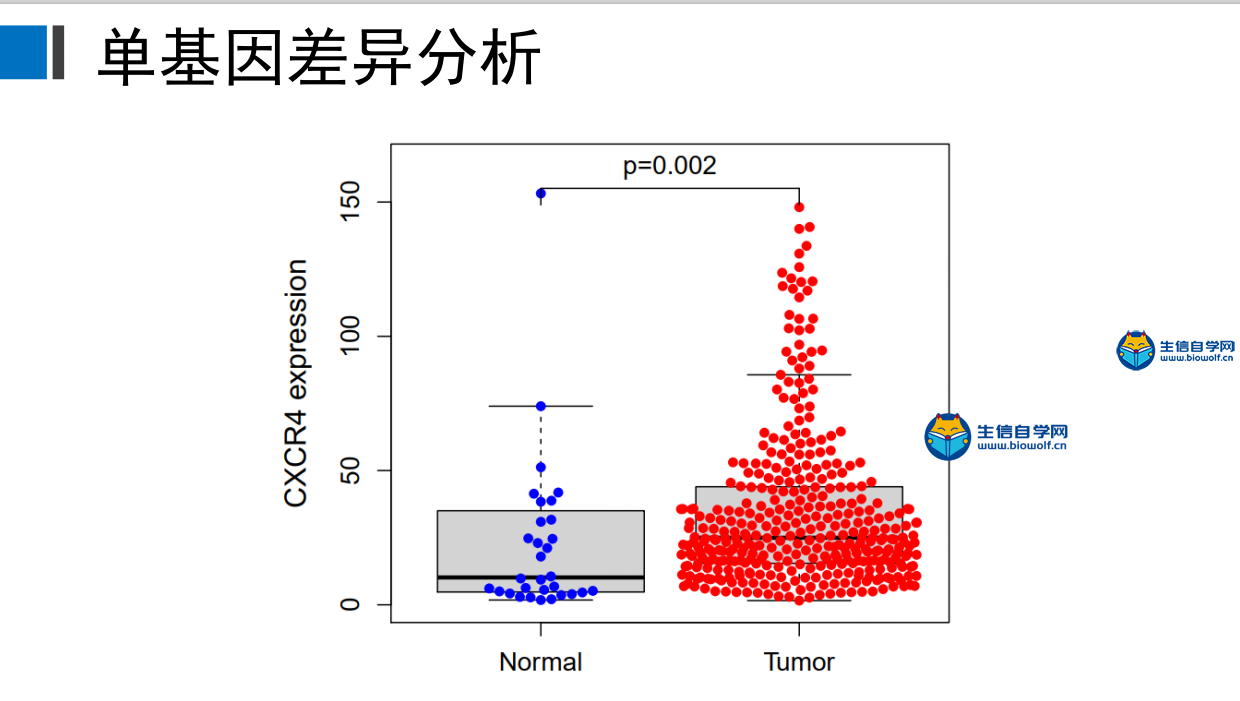 下面我们来绘制这样一个图形,我们首先要准备基因的表达文件,里面包含着基因的表达量,然后是我们的脚本文件。  我们打开R导入我们需要的包,然后将脚本文件拷贝到R中运行,等待运行完成,我们就可以得到我们需要的差异图形。 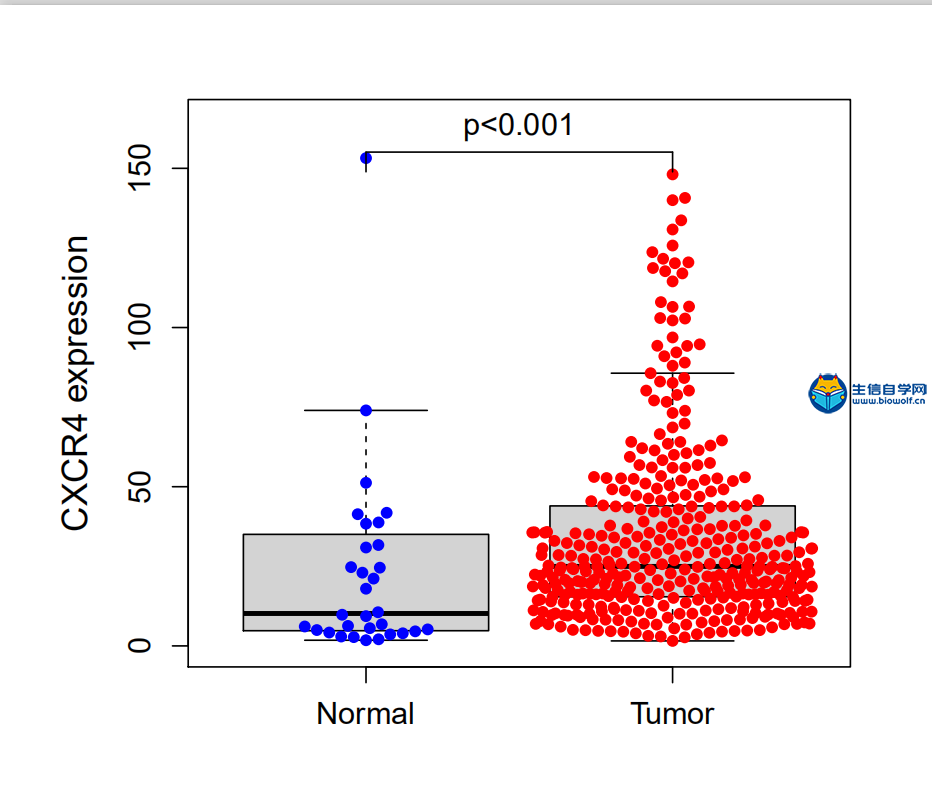 配对差异分析 我们已经比较了我们的目标基因,在正常样品和肿瘤样品里面是否有差异?但是我们在做差异的时候的时候,我们发现TCGA的数据,它的正常样品数目是比较少的,而肿瘤样品数目比较多,所以我们就希望对这个样品进行配对,就可以得到这样一个图形:在这个图形里面我们的样品是一一配对的,每个正常样品它都对应一个肿瘤样品。当然这里的正常样品代表的是癌旁的组织,我们只把这些有对应关系的样品进行差异分析,得到这样的结果。在这个图形里面,它的横坐标是样品的类型,纵坐标是我们选取的基因的表达量,然后我们就可以比较一下这个基因,他在正常样品和肿瘤样品里面是否有差异,同时我们可以看到大对于大多数病人来说,他在正常样品里面的表达量是更低的,因为这个线段从左到右它是往上走的。但是也有几个样品,这几个样品,它对应的肿瘤样品它的表达是更低的,但是从整体的范围来看,从左到右这个表达是上升的,所以我们就可以看出这个基因。他在肿瘤样品里面是上调的。 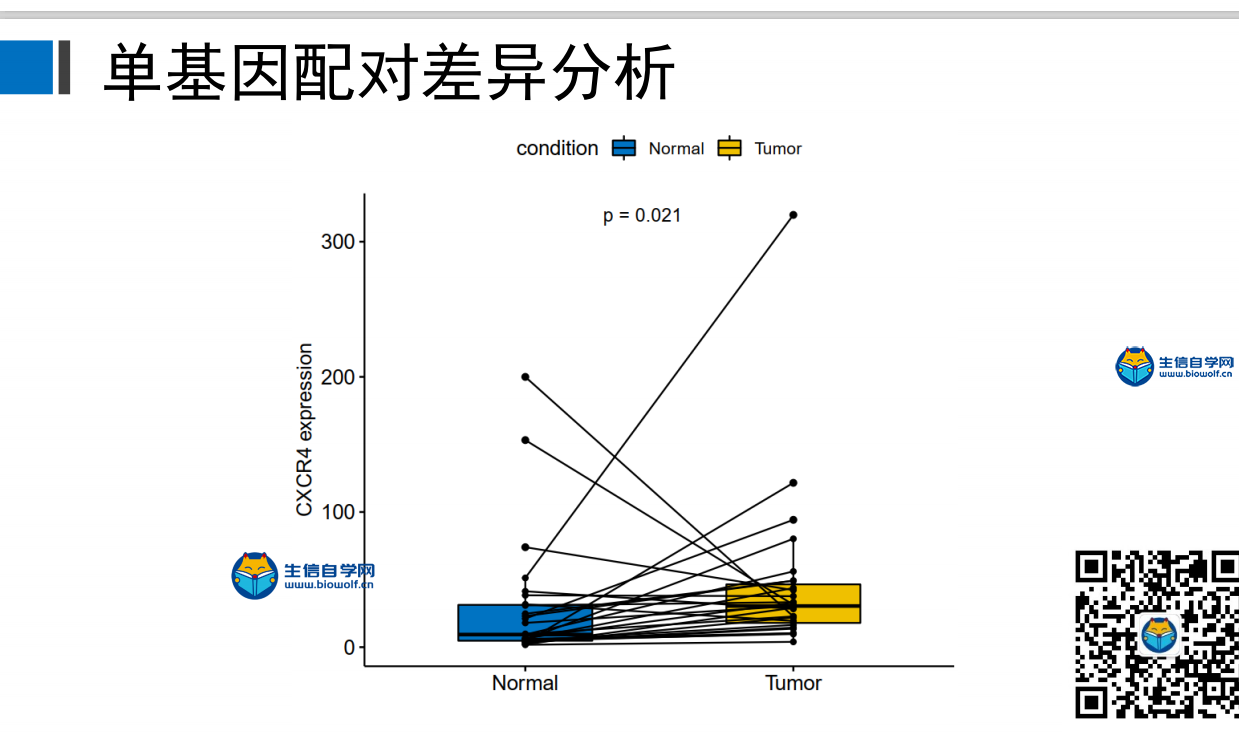 我们做差异配对分析和做差异分析用的输入文件是一样的,唯一不同的是脚本不同,然后我们要在R中运行我们的脚本,结束后,我们可以得到配对的差异结果文件。  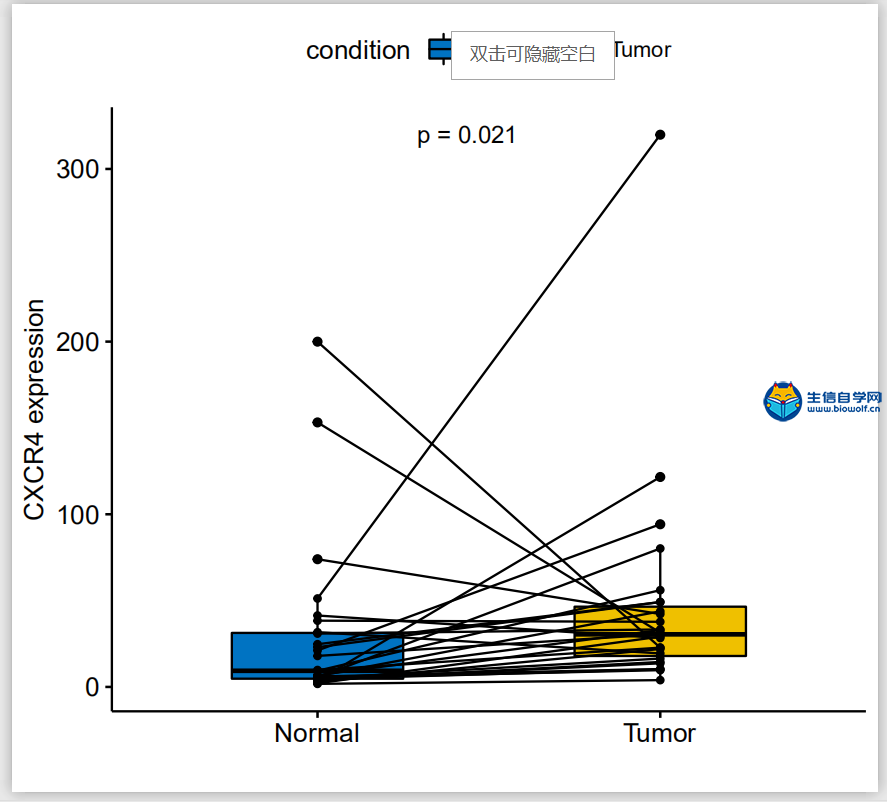 到这里这一篇文章就结束了,下一篇我们要讲解单基因的生存分析和临床相关性分析,感兴趣的学员可以关注我们的公众号查看更多生信咨询。 课程链接: 《TCGA数据库肿瘤微环境视频》 精品课程推荐: 《TCGA肿瘤免疫细胞浸润模式挖掘》 《GEO数据库免疫细胞浸润视频》 《甲基化免疫细胞浸润模式》 《TCGA数据库肿瘤微环境》 《TCGA数据库肿瘤突变负荷》  
责任编辑:伏泽 作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载! |





