TCGA免疫基因对和生存时间合并免疫基因对在上一篇文章中 ,我们已经得到了TCGA数据库和GEO数据库的免疫基因的矩阵,得到免疫基因的矩阵之后我们就可以用免疫基因的矩阵去构建免疫基因对。 下面就是我们构建免疫基因对的结果: 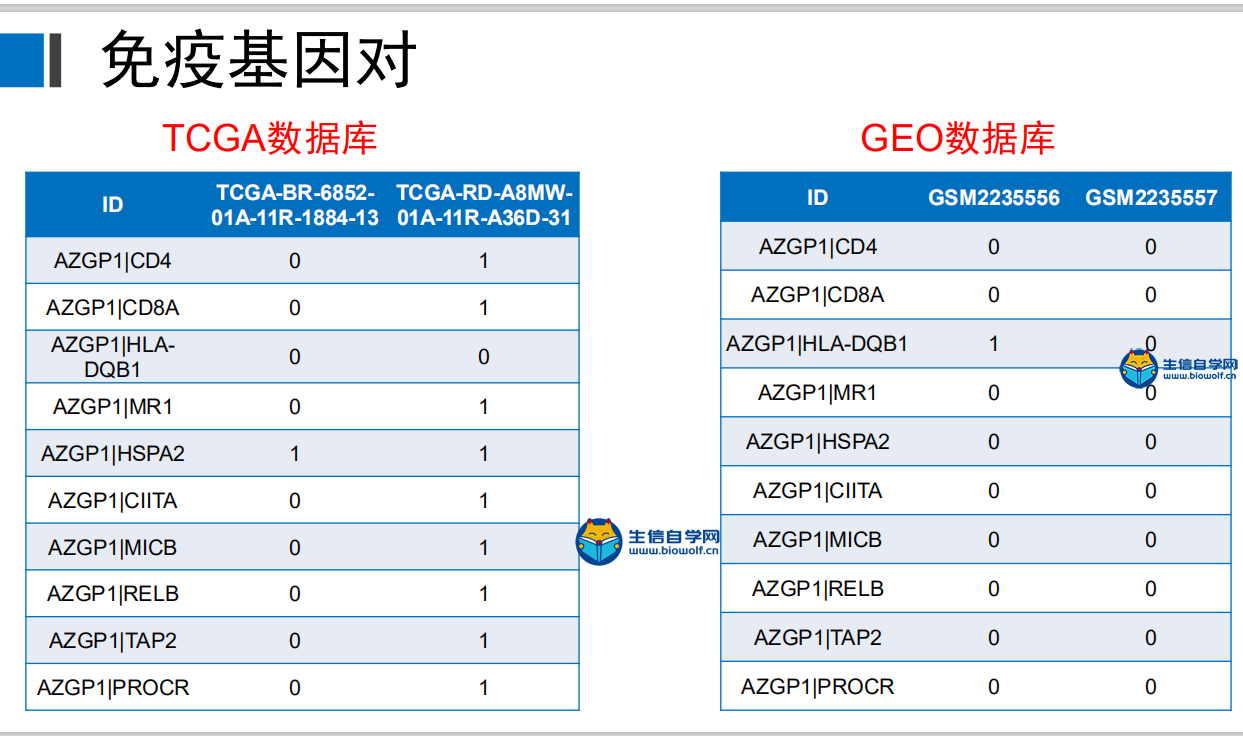 我们首先看下TCGA数据库,它的行名是免疫基因对的名称,列名是样品名,这个矩阵之中的数据只有0和 1,其中的含义以第一行为例,0表示在这个样品中AZGP1基因的表达低于CD4基因,1表示AZGP1基因的表达高于CD4基因。 而GEO数据库也是和TCGA数据库是一样的,这里就不在详述。 下面我们来得到这样的免疫基因对 我们事先准备好输入文件,就是我们上一篇文章提取GEO免疫基因表达得到的两个文件。 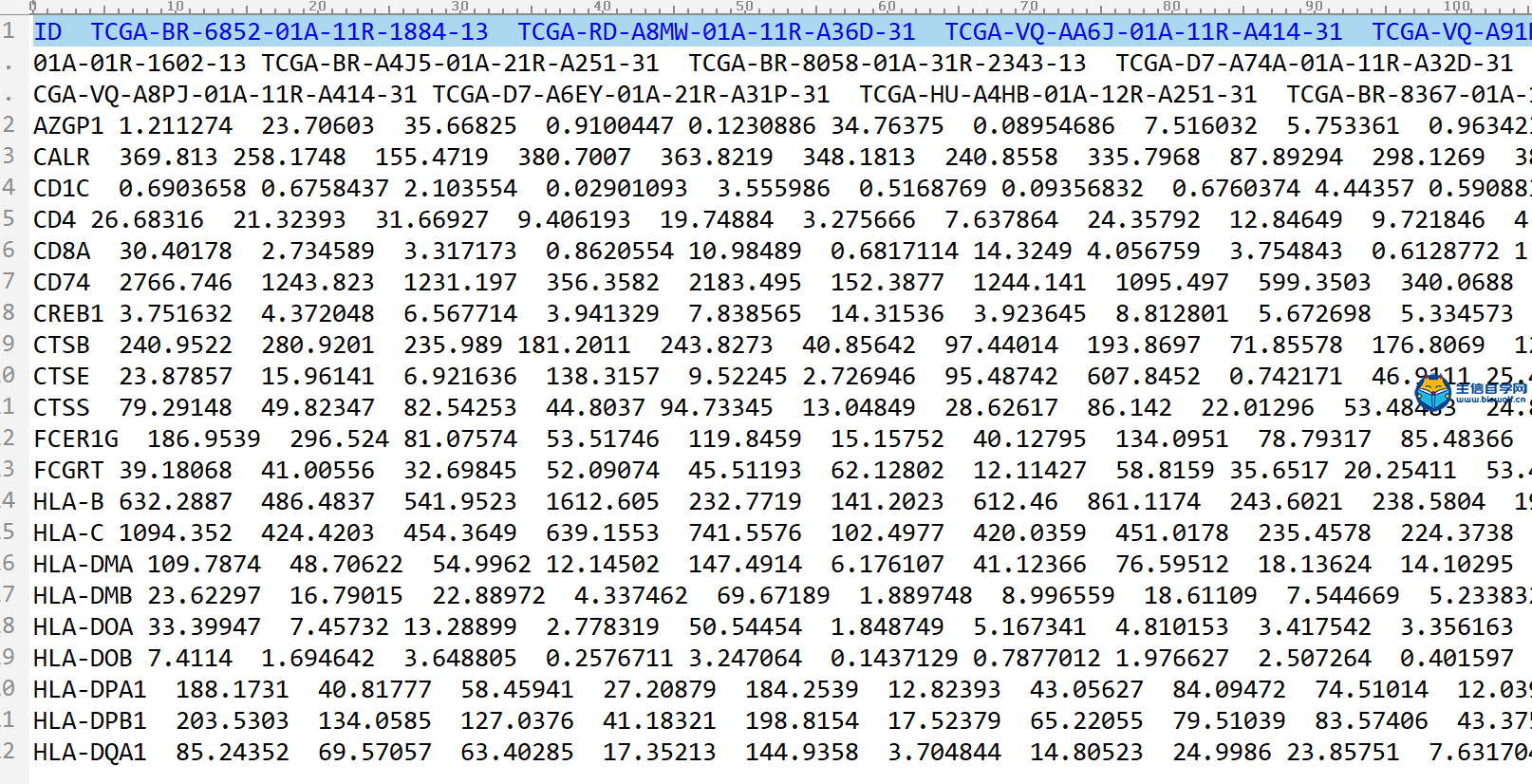 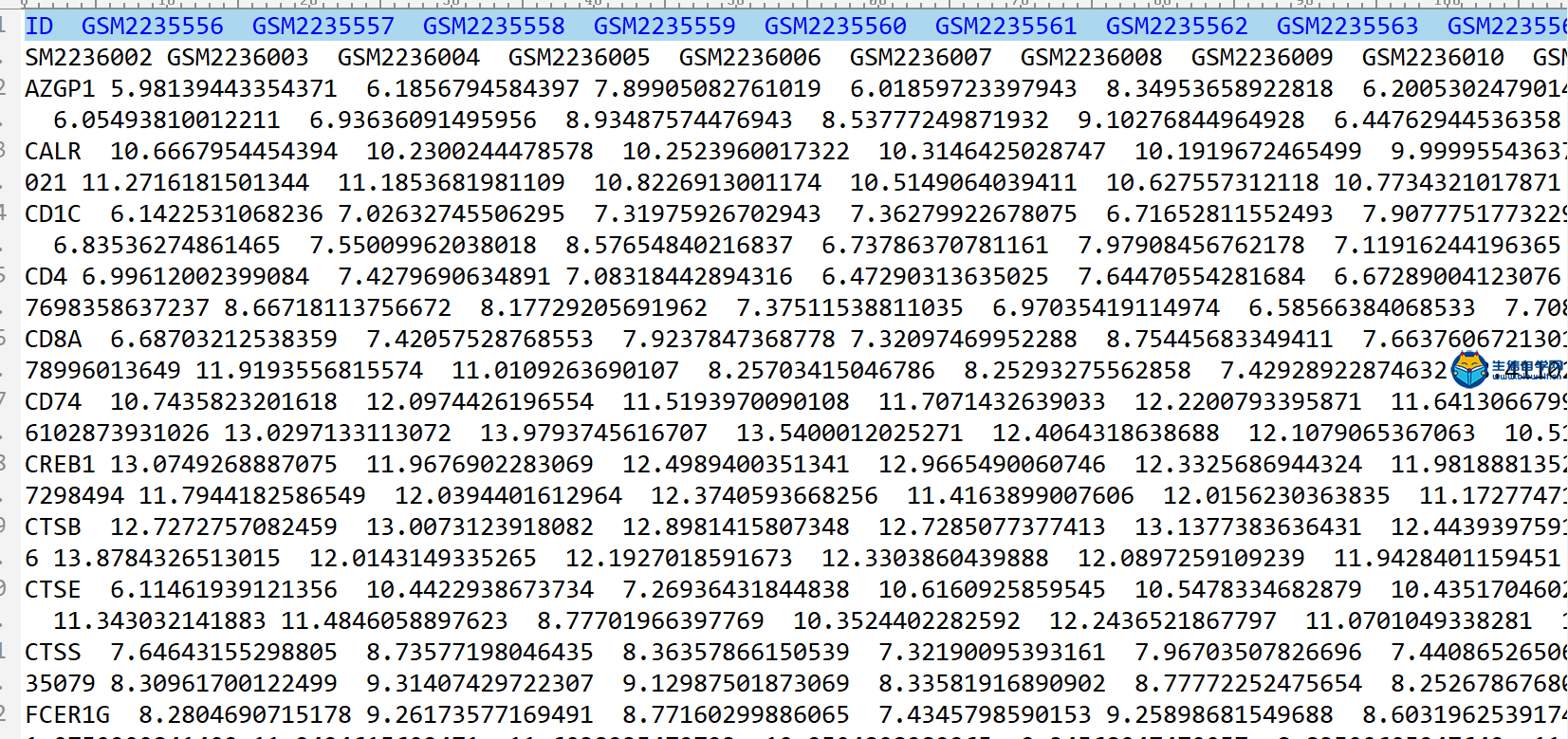 通过R运行我们的脚本文件,我们会得到两个文件,这就是我们免疫基因对的文件。   TCGA免疫基因对和生存时间合并 得到了免疫基因对之后,我们就要将TCGA的免疫基因对和病人的生存时间合并,合并之后我们就可以得到这样的一个矩阵 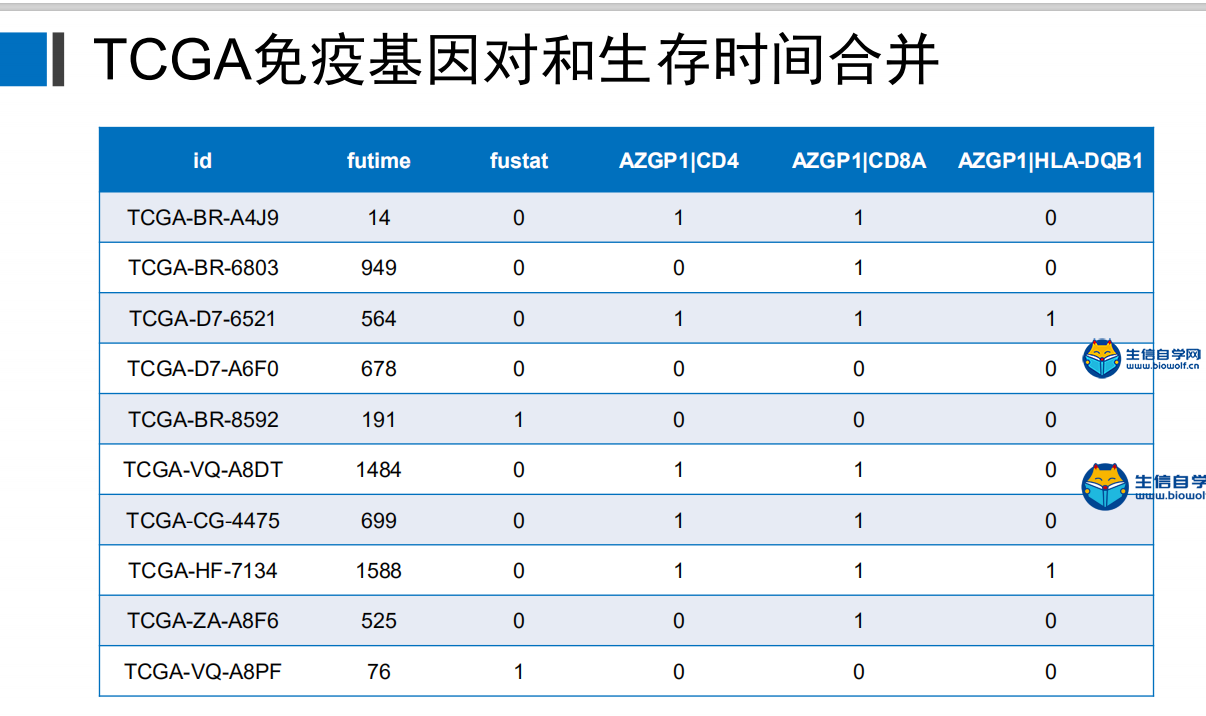 它的第一列就是样品的名称,第二列是生存时间,第三列是生存状态,1代表病人已经死亡,0代表病人还存活,接下来就是所有免疫基因对的状态。 下面进行具体的合并操作,需要用到的文件是TCGA的免疫基因对的文件,还有很早以前我们得到的生存时间和生存状态的表格文件clinical.xls, 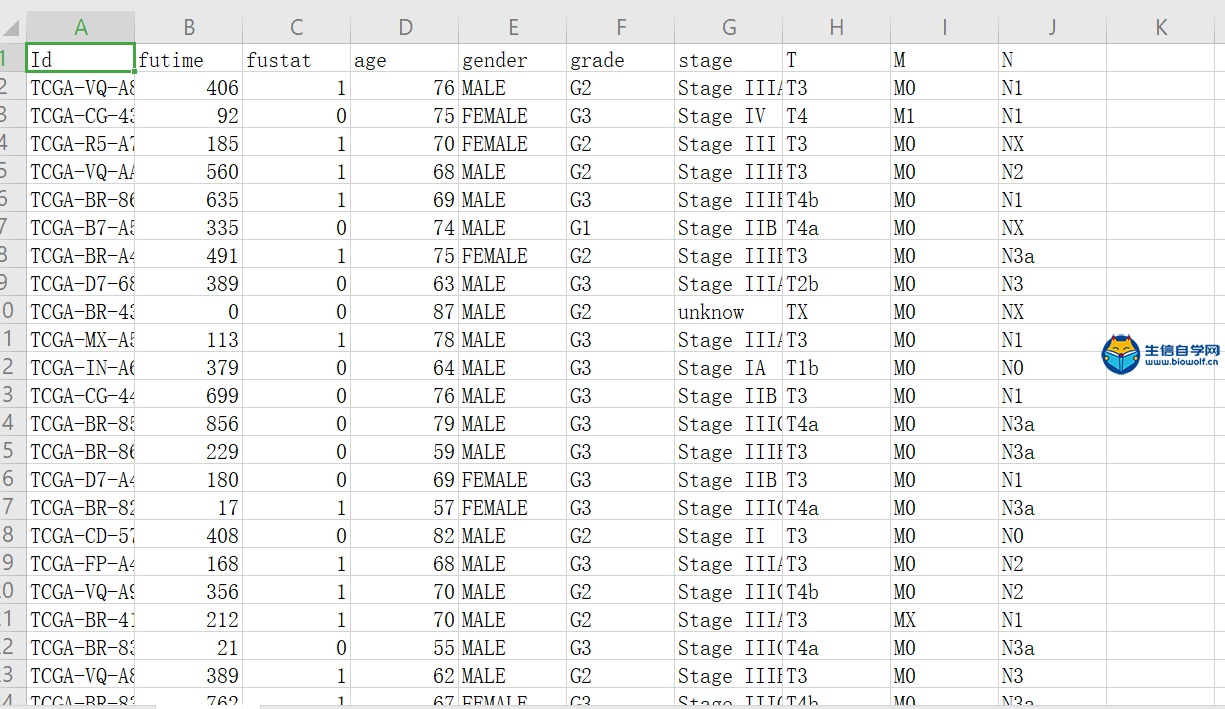 对于生存时间的文件,我们还需要对它进行整理,删除生存时间为空的样品,再删除生存状态为空的样品,最后将样品名,生存时间和生存状态三列单独取出来放在一个txt文件中,这样输入文件就准备好了。 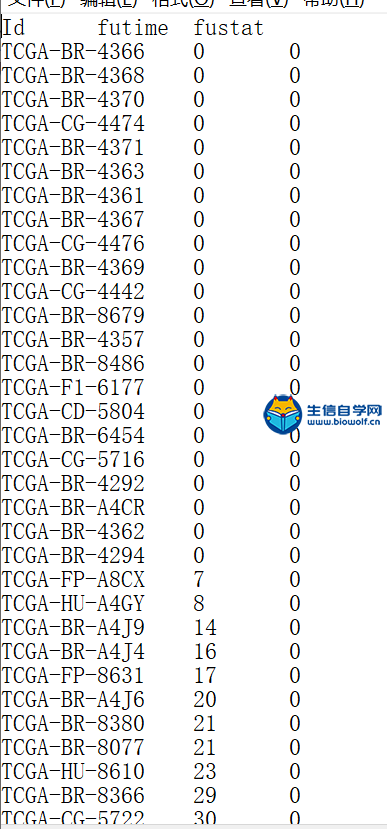 运行我们写好的脚本文件,我们就最终就可以得到免疫基因对和生存时间、生存状态合并后的文件。 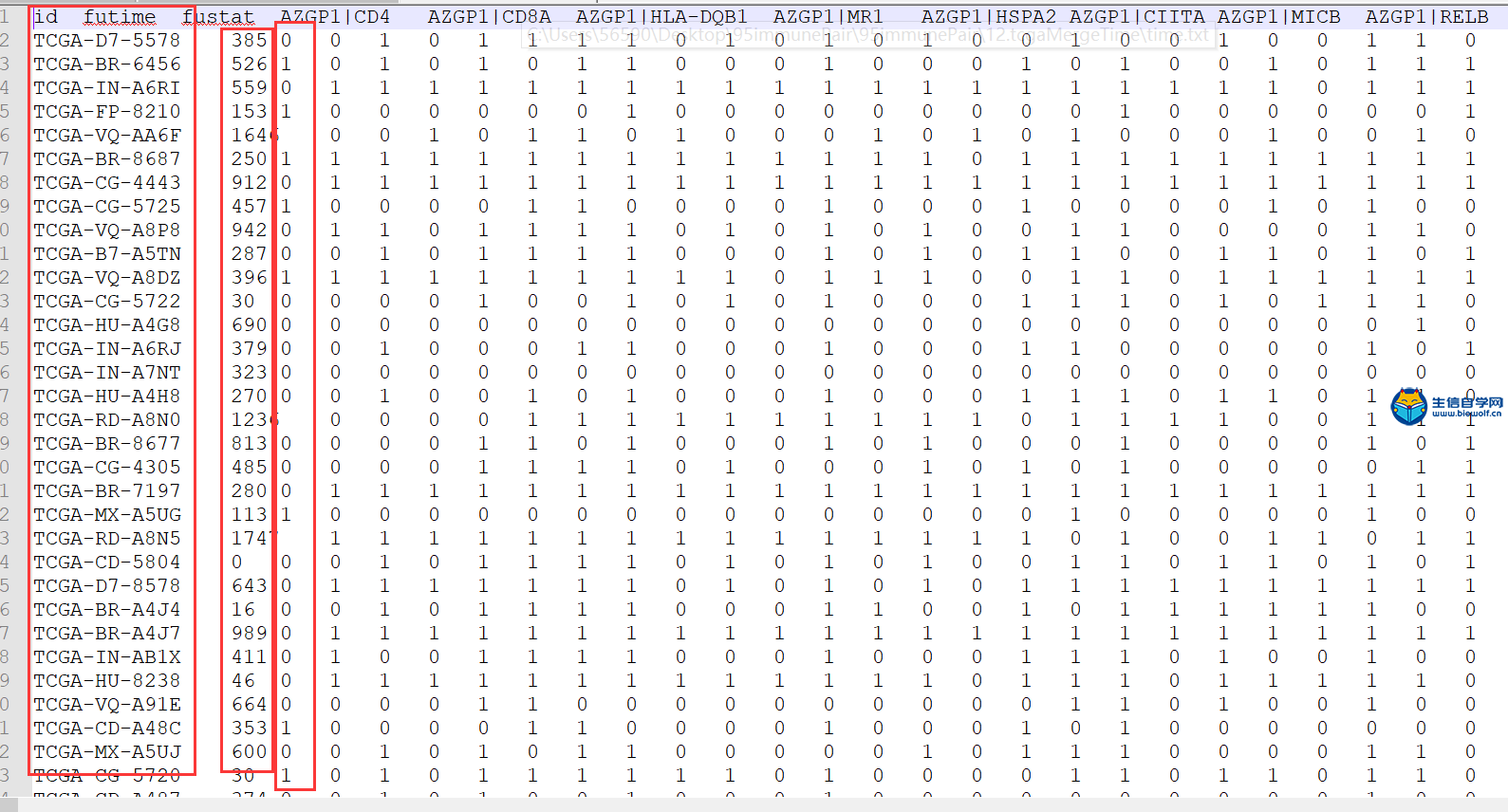 视频购买链接: 《免疫基因对文章套路视频》 精品课程推荐: 《GEO数据库miRNA芯片挖掘》 《甲基化肿瘤分型文章套路视频》 《TCGA肿瘤免疫细胞浸润模式挖掘》 
责任编辑:伏泽 作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载! |





