提取单因素显著的lncRNA单因素cox分析上一篇我们通过脚本对所以基因批量的做了生存分析,接下来我们就要同通过单因素的cox分析得到跟生存相关的一些长非编码RNA,然后对其P值进行排序,选出满足条件的一些长非编码RNA来构建模型。 在构建模型的时候,我们也会采用一些方法,去除基因直接的重复性,比如两个基因如果具有共表达的关系,那这样的基因我们就去除掉。 我们首先要进行的单因素的cox分析,在cox分析结束后,我们会得到这样一个表格,这个表格的第一列是长非编码RNA的长非编码RNA的名字,第二列是HR风险值,如果大于1的话就是高风险的lncRNA,小于1的话就是低风险的lncRNA,通过第三列的z值我们可以得到第四列的Pvalue。 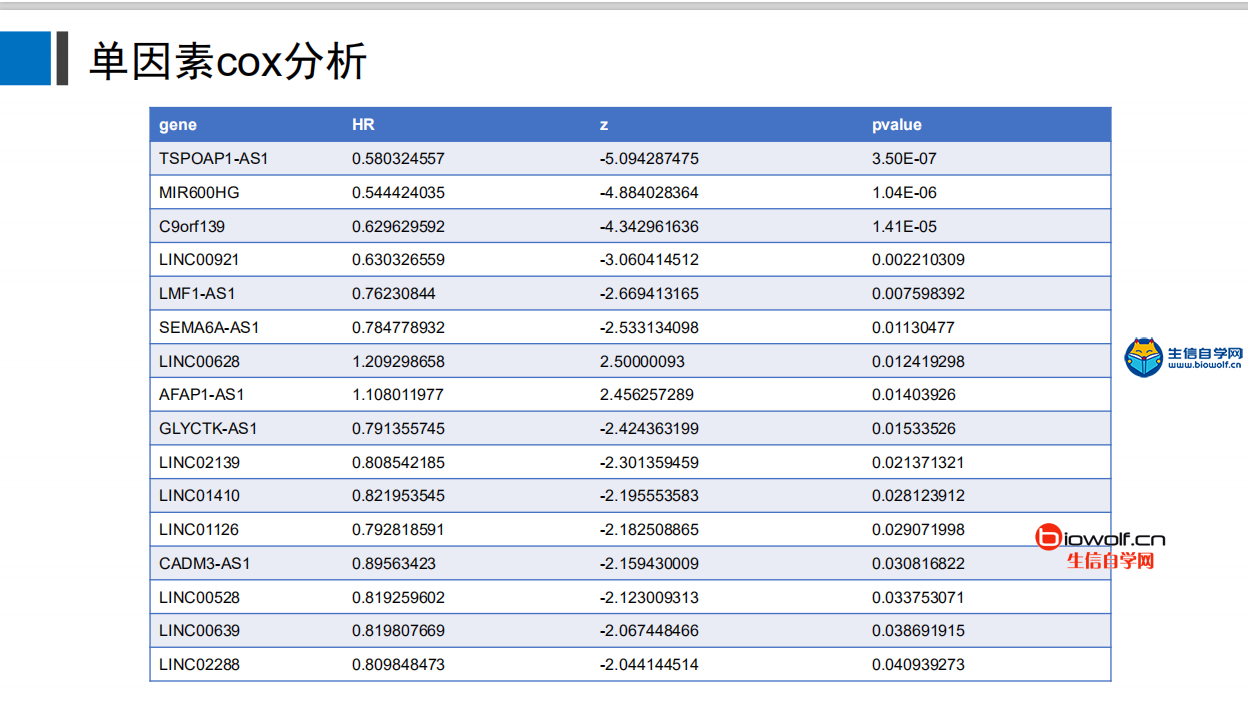 下面我们看一下做单因素分析需要的输入文件 着个文件中就包含样品的id,生存时间,生存状态和长非编码RNA的表达量。有了这些数据,我们就可以做cox分析。 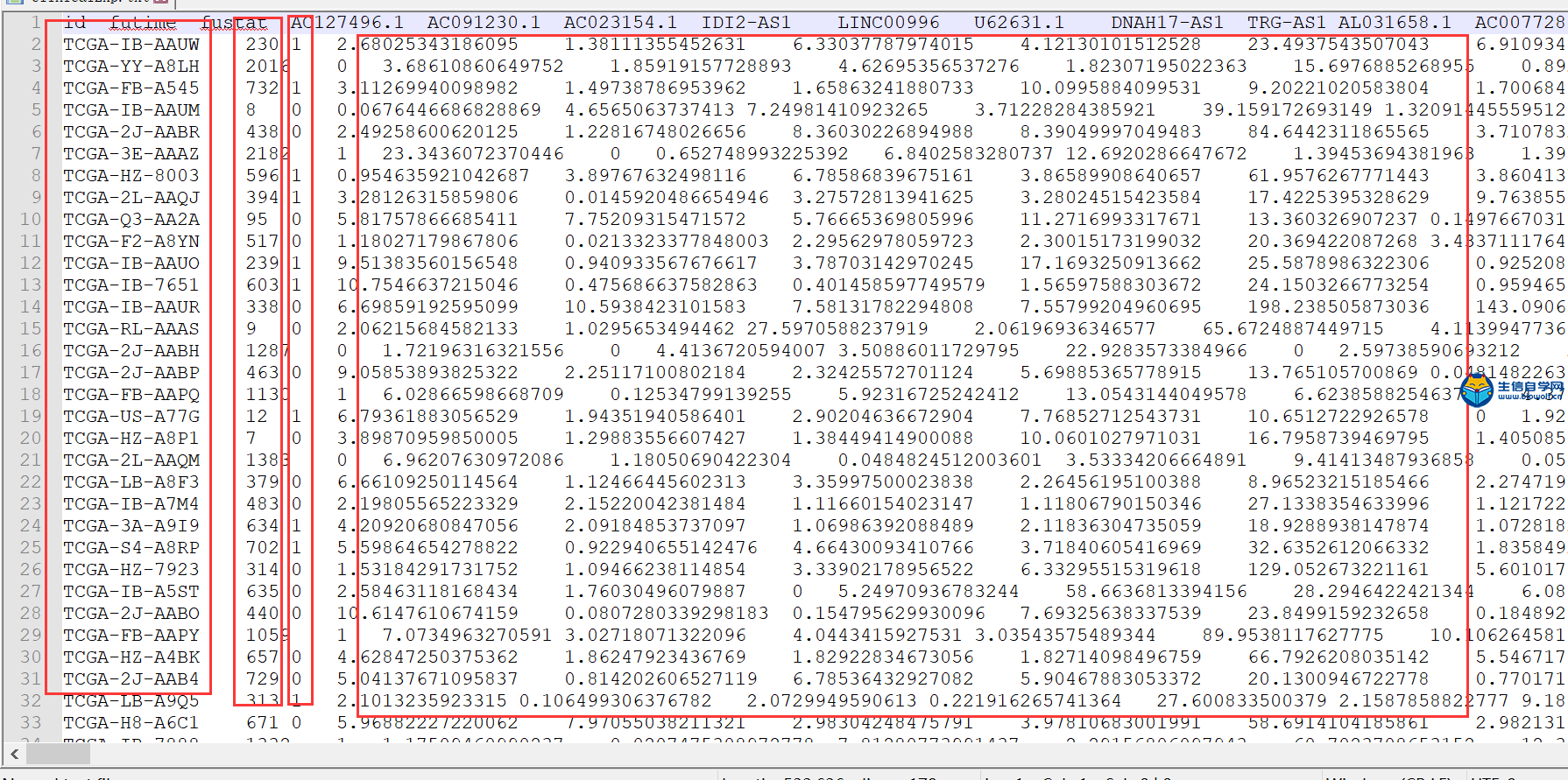 再将我们事先准备好的脚本拷贝到R中运行后,我们会得到一个表格,这就是单因素cox分析后的结果。  提取显著的lncRNA 得到单因素cox分析的结果后,我们要提取出其中显著的长非编码RNA,所以我们要通过脚本,将结果文件的显著RNA找到并在lncRNA的表达量文件中将这些RNA的具体数据提取出来单独放入一个文件中。 我们先在cox分析的表格中通过排序找到p值最高的基因,将基因命拿出来放在一个新的文件夹中,这里我们就找出了7个P值最高的基因。  要提取这些基因数据,我们要用到我们事先写好的pl脚本文件,并通过命令行运行pl脚本,运行结束后,我们就得到了七个长非编码RNA的表达量文件,这样我们就可以做后续的多因素的cox分析 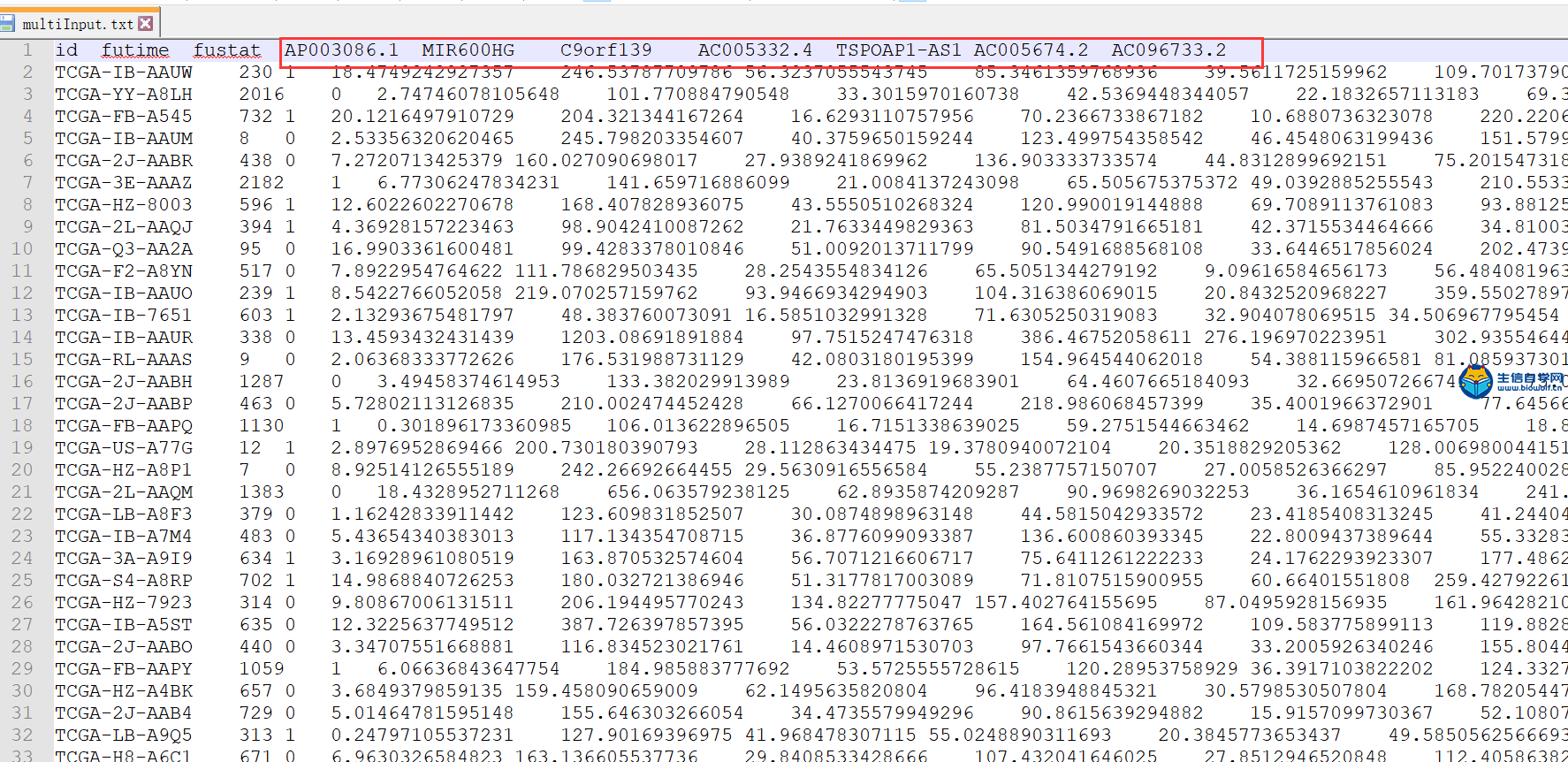 课程链接: TCGA数据库批量挖掘lncRNA视频 相关课程: 长非编码RNA芯片数据挖掘视频 
责任编辑:伏泽 作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载! |





