单细胞测序数据整理PCA TSNE聚类得到marker基因
时间:2019-07-30 来源:生信自学网 作者:乐伟
微信公众号:biowolf_cn 点击:次
| 前面我们给大家介绍了如何从GEO数据库下载单细胞测序(scRNA)的数据,接下来,生信自学网给大家简单重现一下数据整理以及后续的分析: 1、把数据解压,在excel里面简单整理,就可以得到行名是基因名,列名是样品名的矩阵,“PT089_P1_A01“中PT089代表样品的名称,P1_A01代表细胞编码; 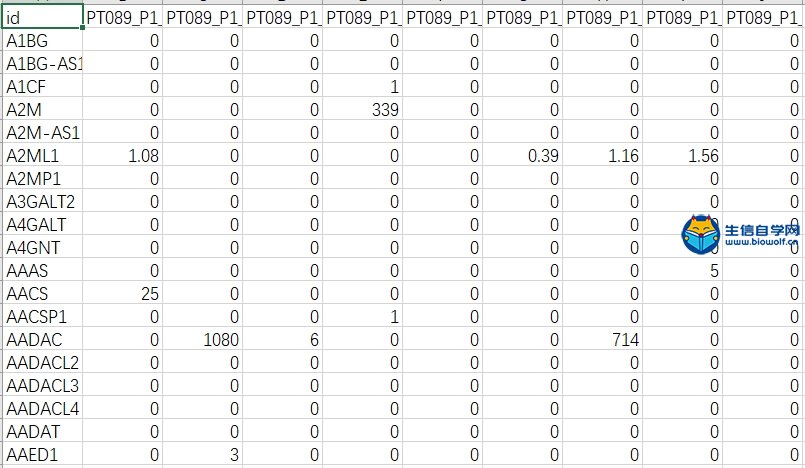 如果需要学习整个单细胞测序数据分析过程,可以学习生信自学网给大家准备的课程: 《单细胞测序数据分析视频课程》 2、得到矩阵之后,我们需要对数据进行质控和数据过滤,在图形左1里面,横坐标代表样品名,纵坐标基因的数目;左2图横坐标代表样品,纵坐标代表测到所有基因的序列数目;左3代表线粒体数目,因为这组数据没有相关数据,所以都是0。 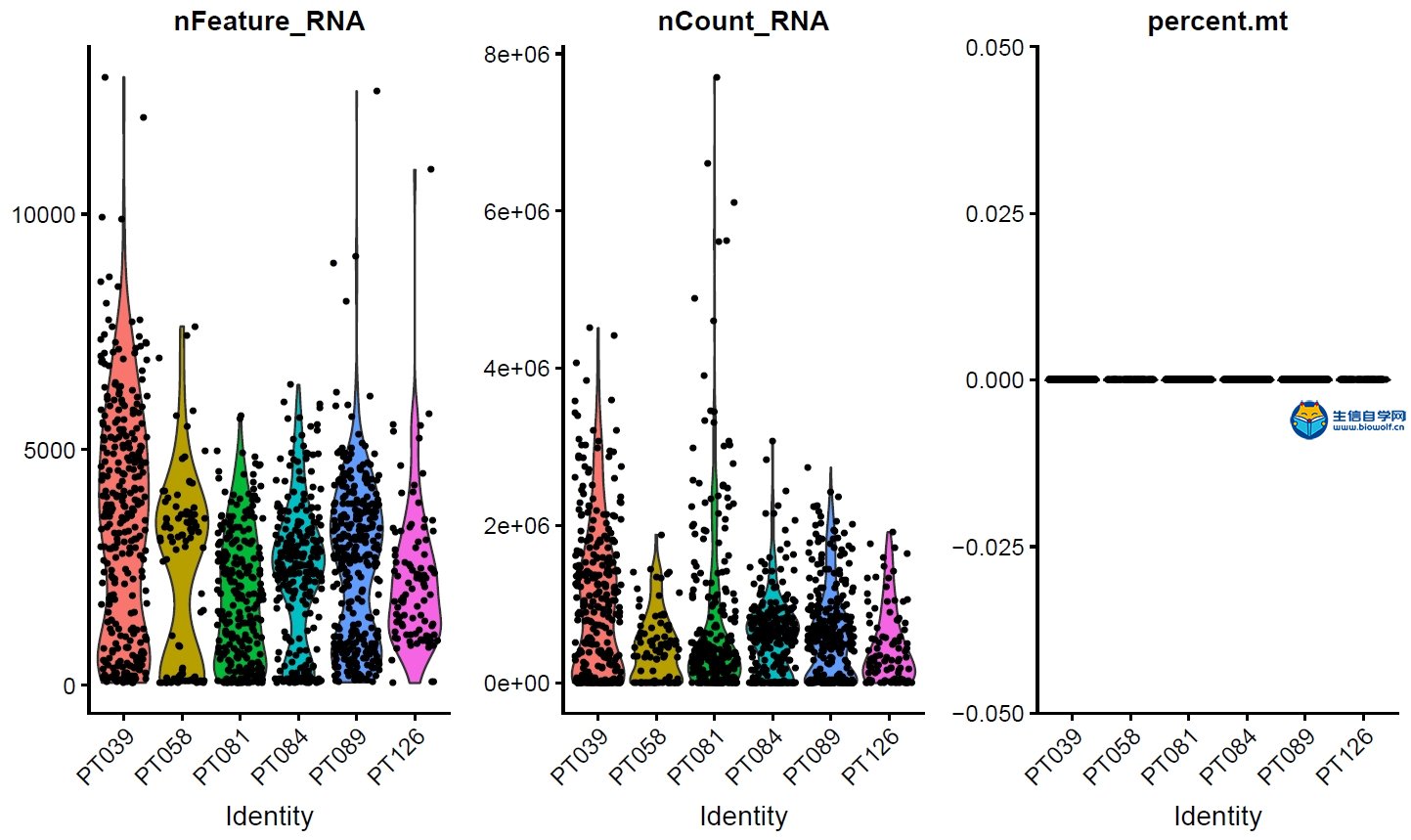 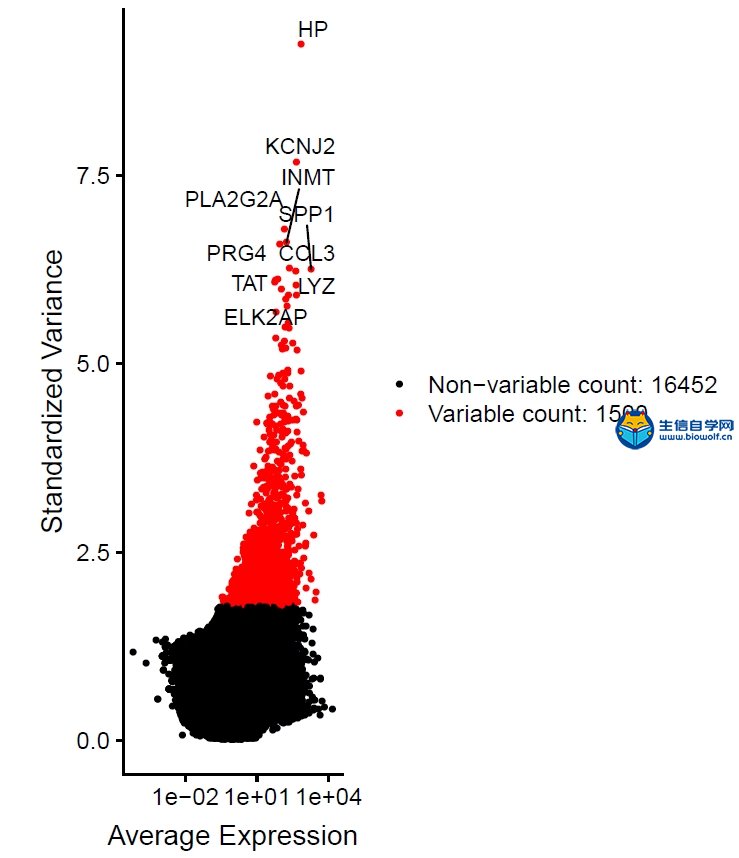 3、主成分分析,每个点代表一个细胞,不同颜色代表不同的样本;接下来我们可以做PCA的热图,跟每个PC相关的基因表达谱情况。 每根曲线代表一个PC,每一个PC可以理解为一部分基因的集合,前面得到的1500多个基因就得到20个PC,图形里面每个PC后面有个P值,这里的P值是指实际PC里面的关键基因和理论PC可能存在基因的相似程度,P值越小,说明PC实际基因和理论相似程度高。  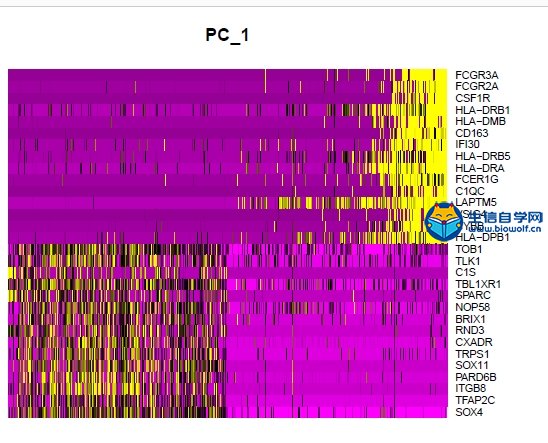  4、TSNE聚类分析 上一步选好了PC,接下来做TSNE聚类分析,在图形里面,聚成了15类,也就是15个Cluster,然后可以得到每个样品的细胞是属于哪个Cluster。绘制Cluster的聚类热图,可以根据热图得到每个聚类的marker基因。 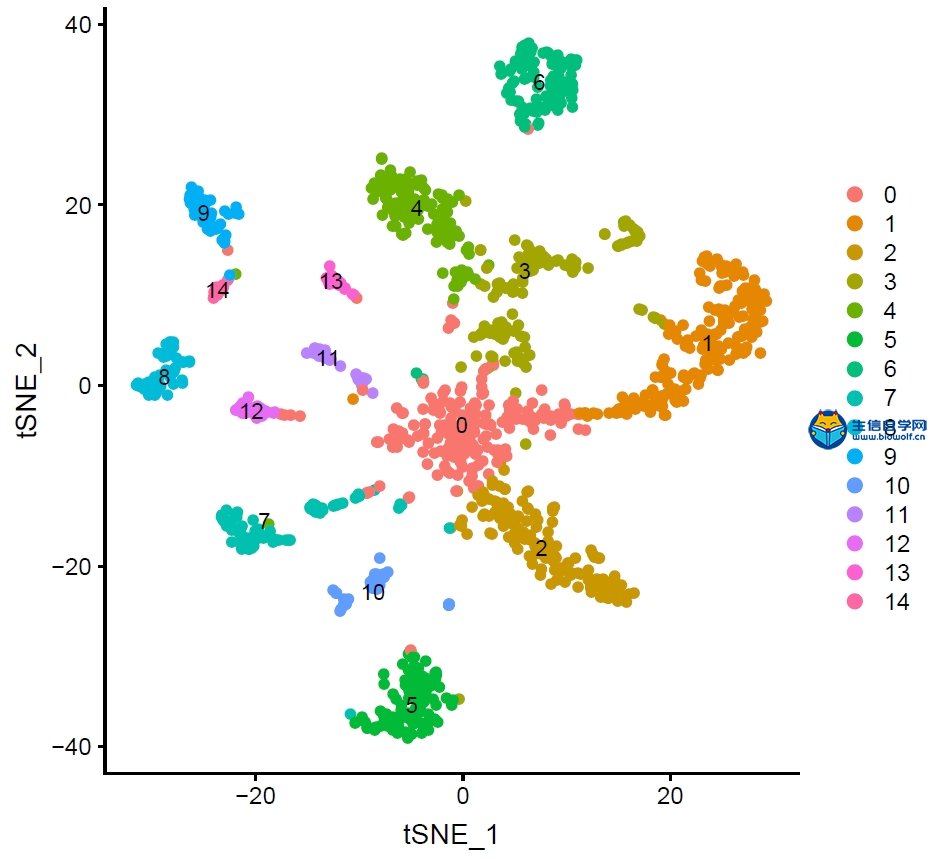 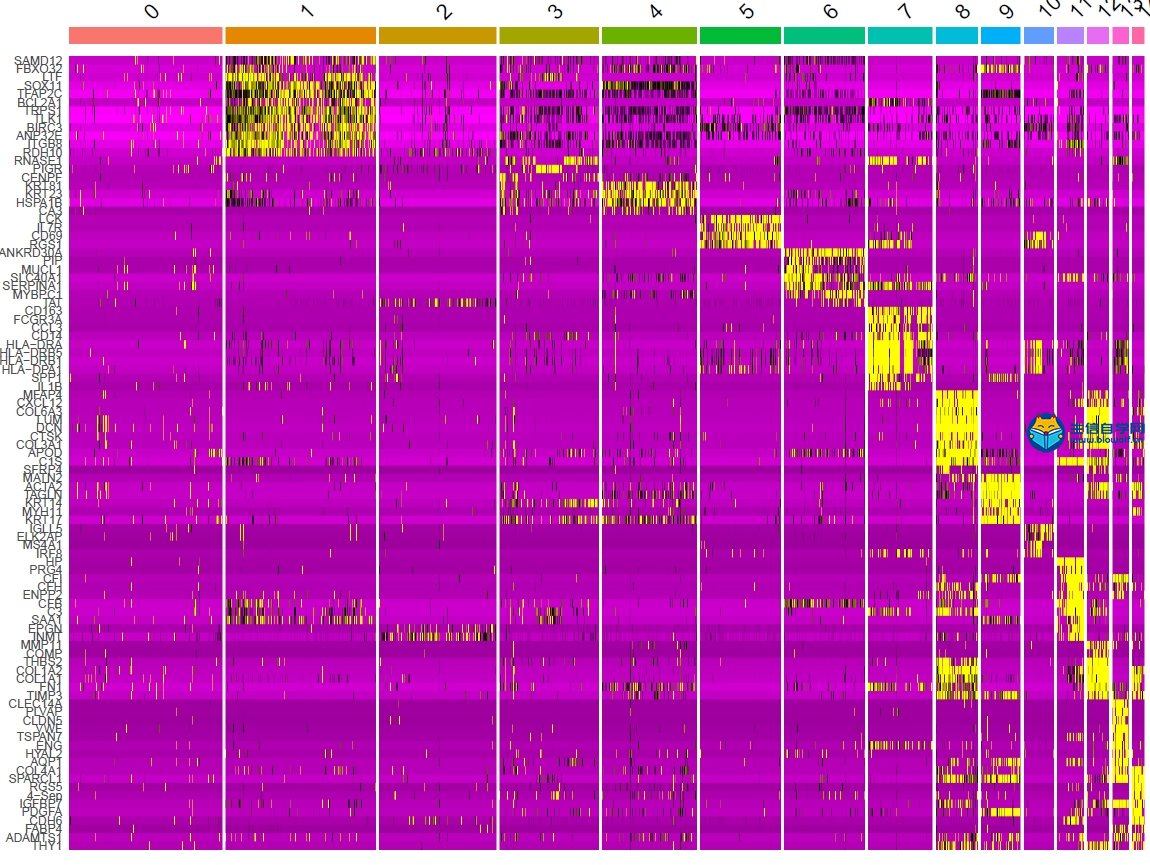 5、marker 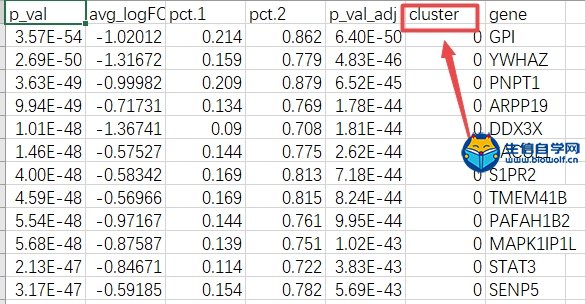 如果需要学习整个单细胞测序数据分析过程,可以学习生信自学网给大家准备的课程: 《单细胞测序数据分析视频课程》 (责任编辑:伏泽 微信:18520221056) 
|