基质细胞与免疫细胞的分组差异分析
时间:2020-08-06 来源:生信自学网 作者:刘鑫
微信公众号:biowolf_cn 点击:次
肿瘤微环境联合免疫细胞浸润基质细胞与免疫细胞的分组差异分析1、基质细胞分组差异分析这次我们来讲解一下基质细胞的分组差异分析,我们得到肿瘤微环境的结果之后,我们就可以得到每个细胞基质细胞的打分和免疫细胞的打分,我们就可以根据基质细胞打分的中位值将病人分成高低打分两组,之后我们就可以比较哪些基因在这两组之间是具有差异的,然后我们就可以做差异分析。 做完基质细胞的差异分析后,我们可以得到如下一个表格和一个图形,在这个表格里面,他的第一列是基因的名称,第二列和第三列分别是基因在低打分组和高打分组的均值,第四列是基因的logFC值,如果这个值大于1,则说明基因在高打分组是上调的,小于1,则在高打分组是下调的。然后第五列是基因的P值,对P值进行校正,我们就得到了fdr值。 接着我们来一下如下热图,这个热图它的横坐标是样品的名称,纵坐标是基因的名称,对于样品的话我们可以分为两组,左边是低得分组,右边是高得分组。然后我们就可以比较这些基因在高低得分组中是否具有差异。  我们要做差异分析,我们首先要准备基因的表达矩阵symbol.txt文件,然后还有基质细胞的打分文件scores.txt 。准备好输入文件后,接下来就是我们的脚本文件,脚本文件由生信自学网提供,如果学员感兴趣或是有需要的话可以通过下方链接购买我们的课程。  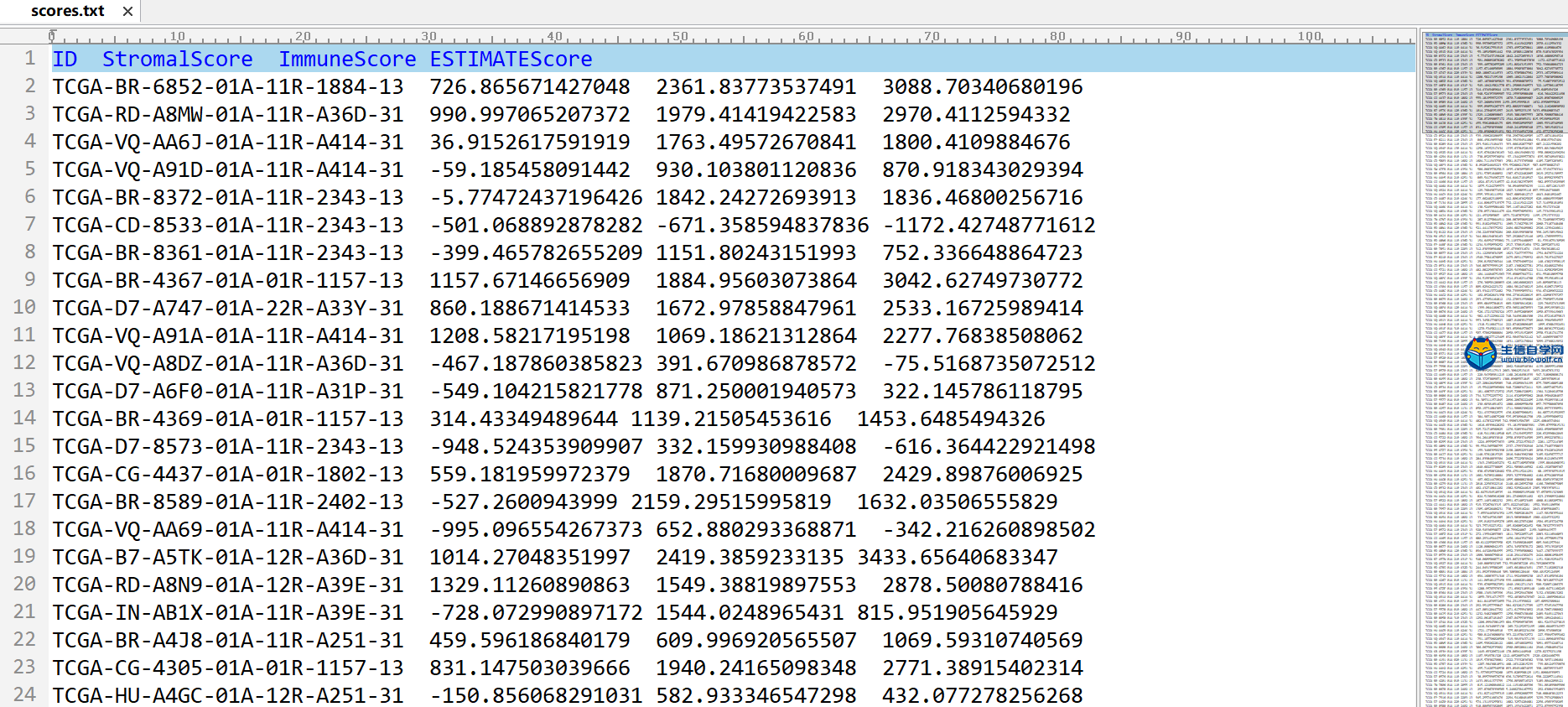 将脚本文件里的脚本拷贝到R中 运行,等待运行完成,我们会得到5个输出文件,分别是所有基因的差异表格文件和文本文件,差异基因的差异表格文件和文本文件,以及热图。  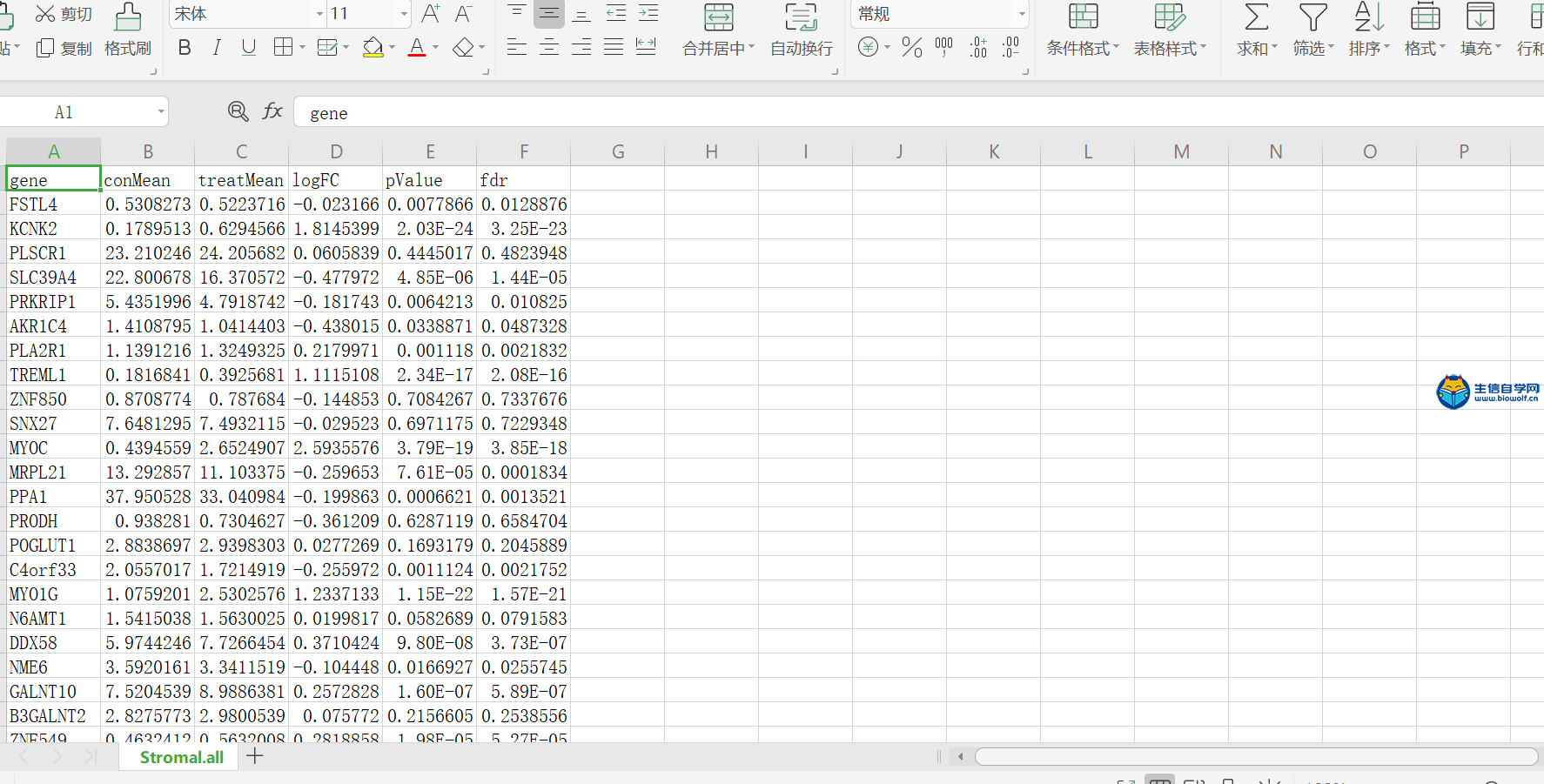 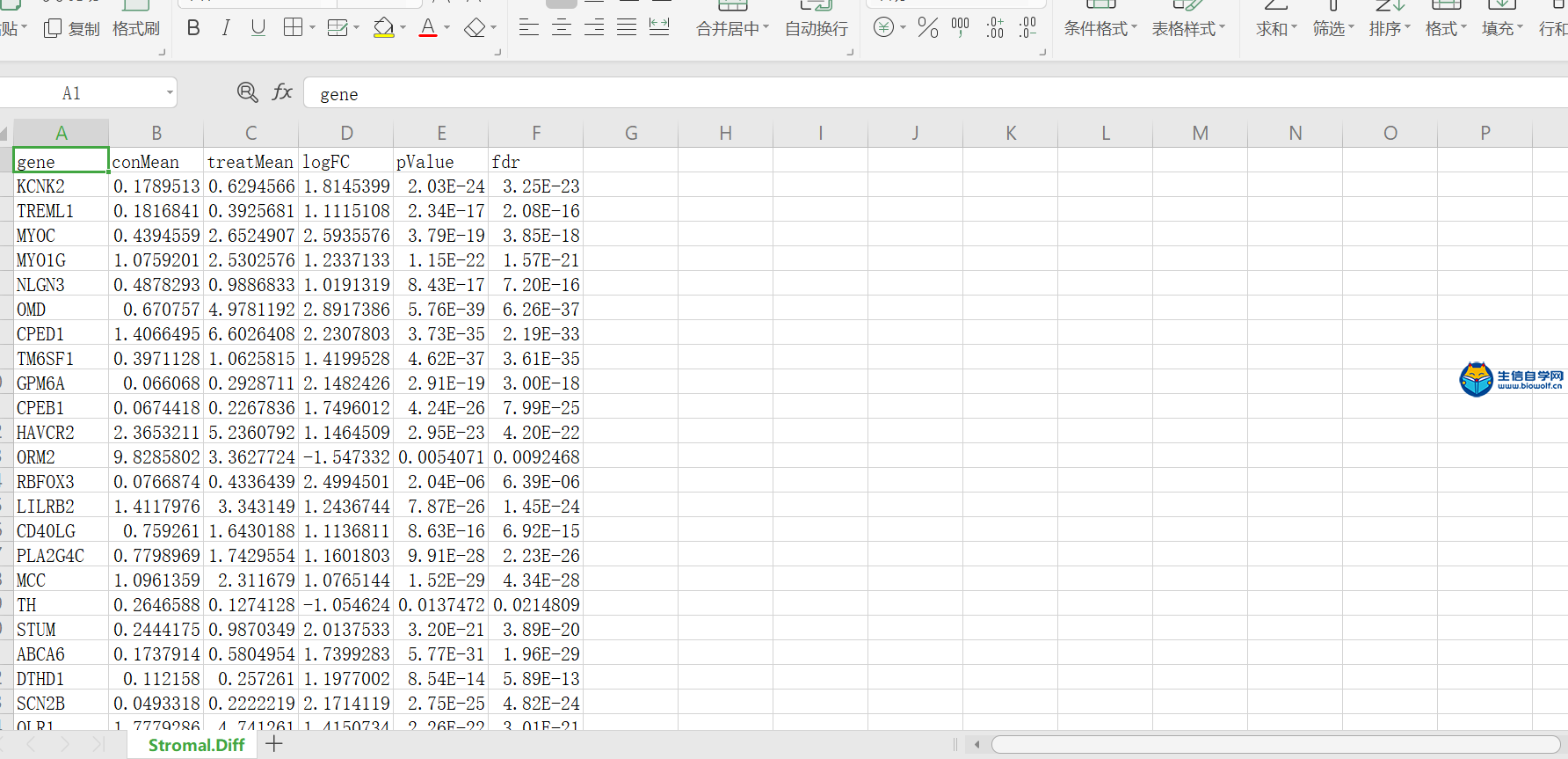  2、免疫细胞的分组差异分析 做完基质细胞的分组差异分析后,我们接下来要做的是免疫细胞的分组差异分析。同样我们会得到如下表格和热图,这里就不再详述。 我们需要准备的输入文件与我们做基质细胞差异分析的文件是一样的,唯一不同的是脚本文件。 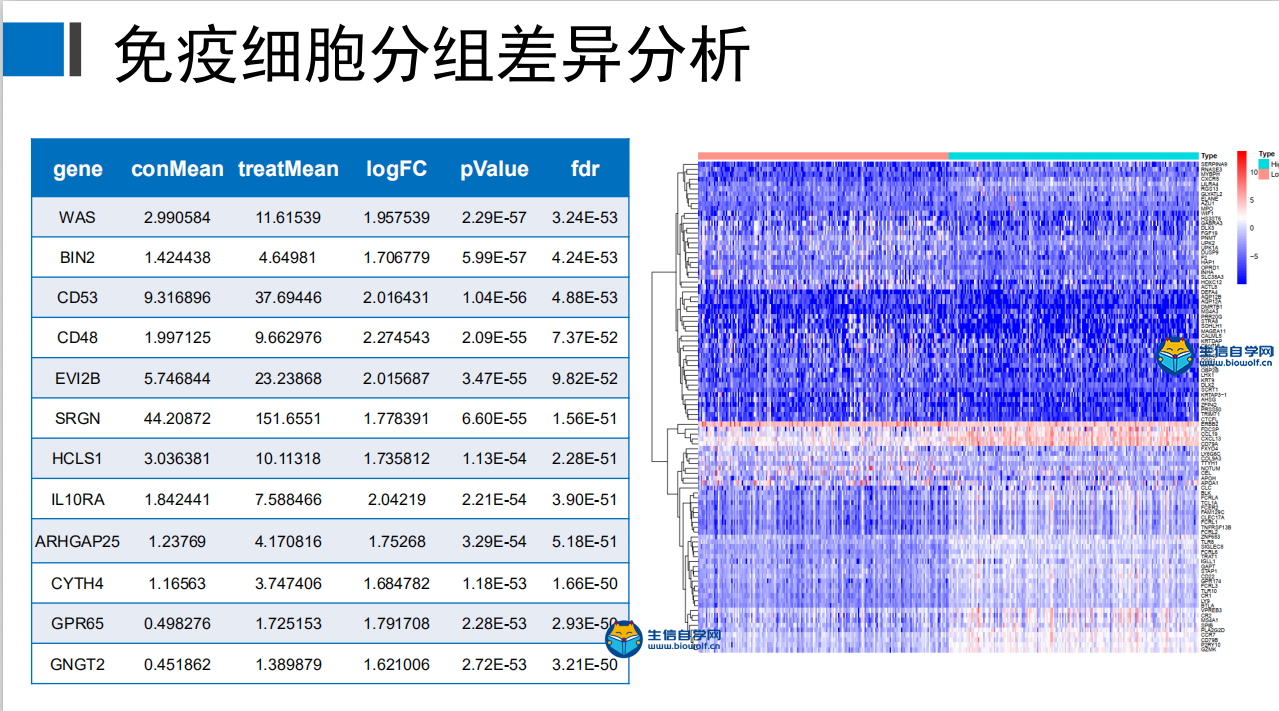 同样运行我们的脚本文件,我们也就得到了5个输入文件,这就是免疫细胞的差异分析的结果文件。  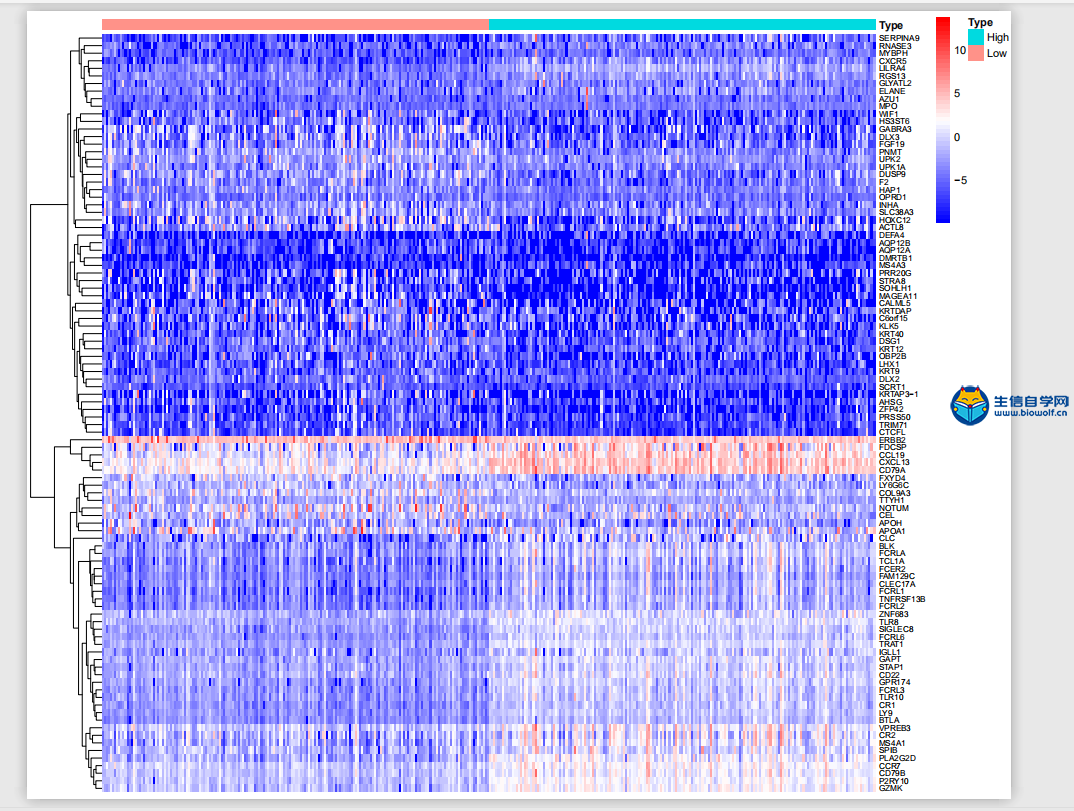 课程链接: 《TCGA数据库肿瘤微环境视频》 精品课程推荐: 《TCGA肿瘤免疫细胞浸润模式挖掘》 《GEO数据库免疫细胞浸润视频》 《甲基化免疫细胞浸润模式》 《TCGA数据库肿瘤微环境》 《TCGA数据库肿瘤突变负荷》  (责任编辑:伏泽 微信:18520221056) 
|
- 上一篇:肿瘤微环境联合免疫细胞浸润微环境生存分析与
- 下一篇:差异Venn图与id转化