TCGA下载和提取临床数据
时间:2018-01-27 来源:原创 作者:BioWolf
微信公众号:biowolf_cn 点击:次
| 一、数据库:TCGA 二、内容:下载临床数据,提取临床数据 三、癌症数据:宫颈鳞状细胞癌CESC 四、方法: 1、可视化下载XML原始文件 2、perl脚本提取XML文件的临床信息,得到临床数据 五、步骤 1、登陆TCGA数据库官方网站,https://cancergenome.nih.gov/ ,点击"Launch Data Portal"进入数据库页面,或者直接登陆数据库网站:https://portal.gdc.cancer.gov/ 。进到数据库网站,点击“Data”,进入可视化选择页面。  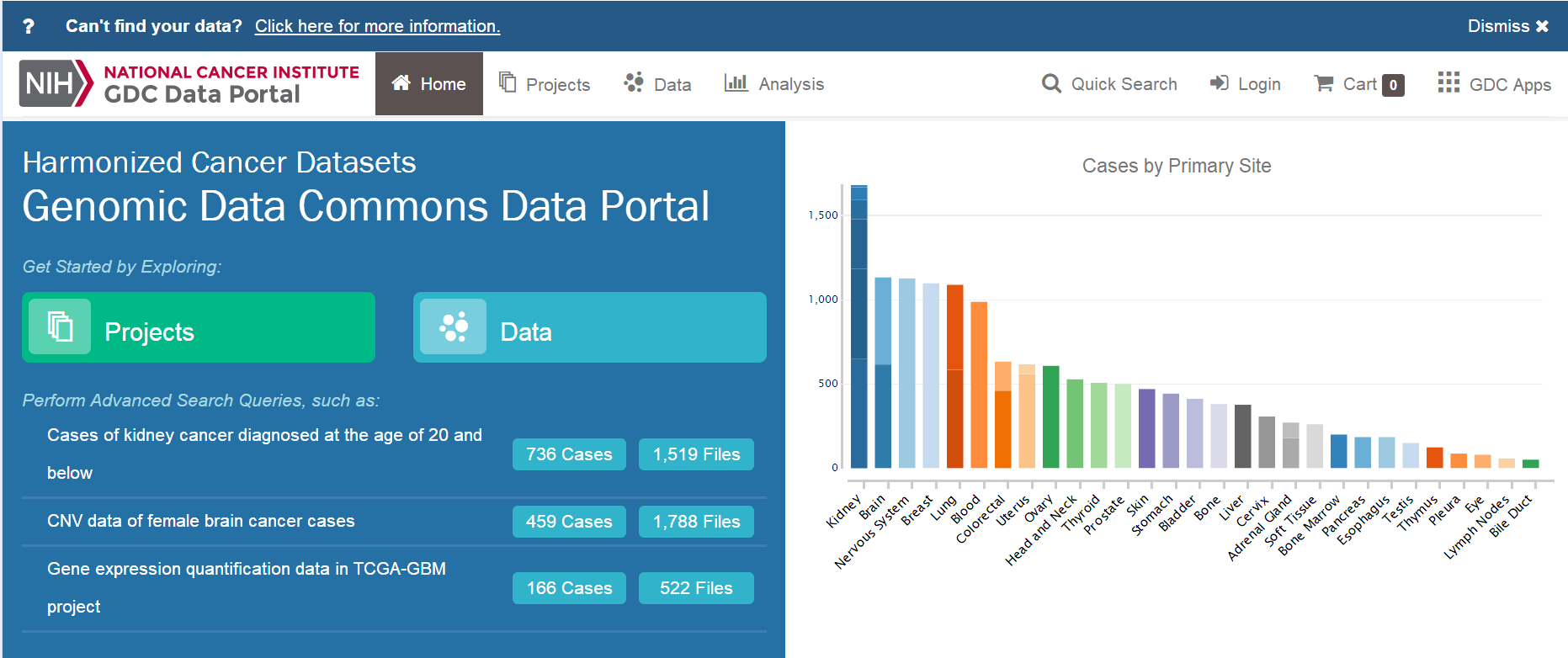 2、选择的方法:CASE选项框依次选择——"Primary Site"-Cervix——"Cancer Program"-TCGA——"Project"-TCGA-CESC——其他默认即可 Files选项框依次选择——"Data Category"-Clinical——其他默认即可 这是右边可以得到Cases数目307个,Files数目307个,大小是20.04M 说明:Case是样本的数据,Files是文件数目,在mRNA的数据时,经常出现Cases的数目和Files的数目是不相等的,这是因为,一个样本可能有多份数据。 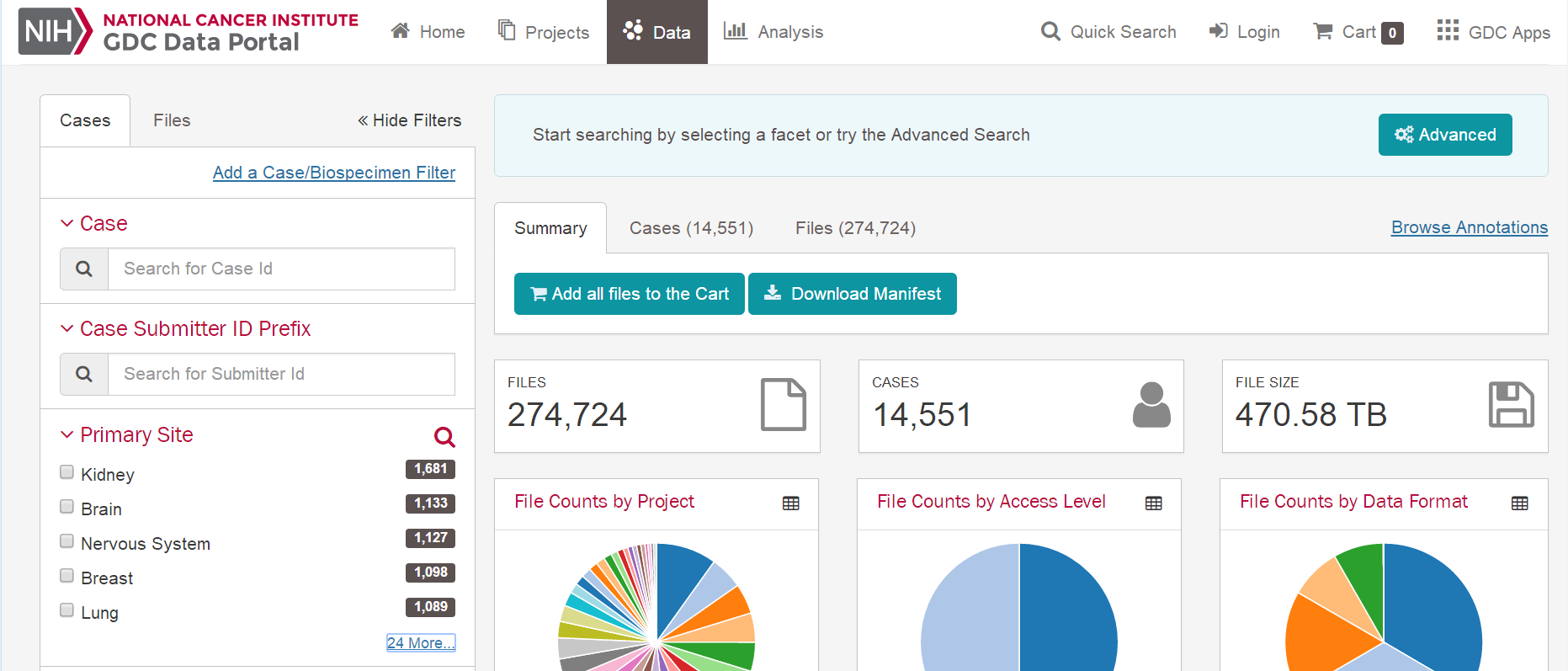
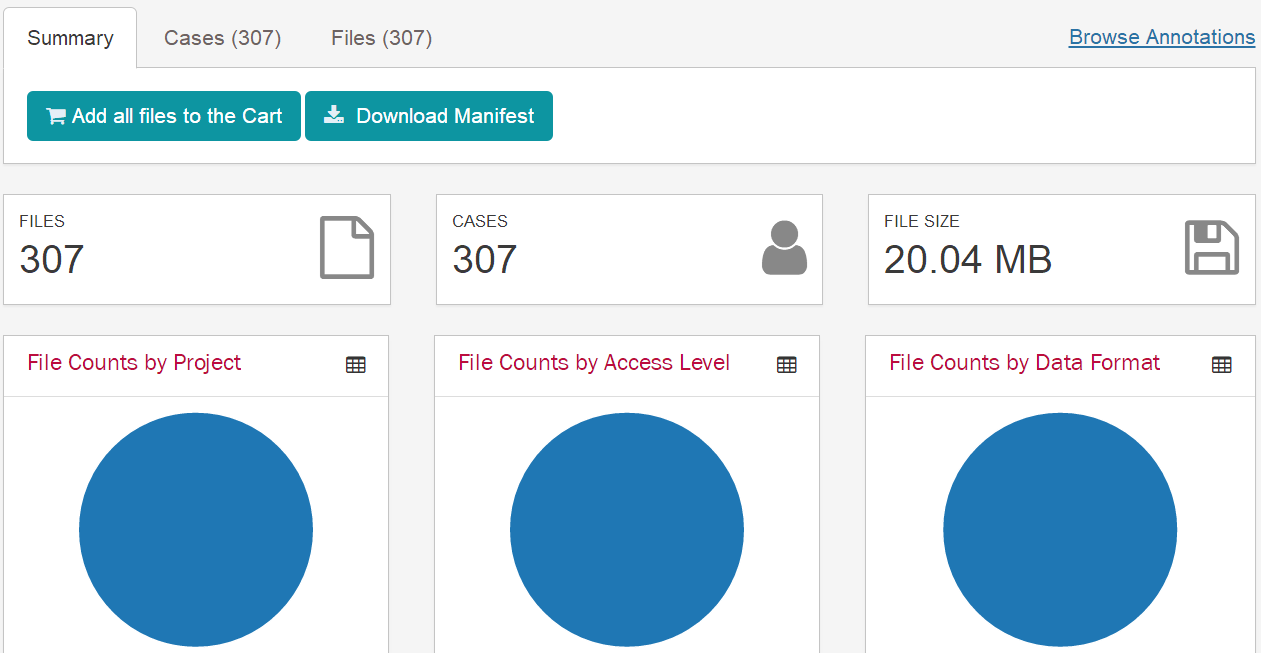 3、点击"Add all files to the cart",然后进入右上角的"Cart"进入数据展示和下载页面 说明:"Cart"是TCGA数据库类似购物车的一个工具,里面是我们选到的数据界面。 4、在“Cart"页面中,我们需要下载3个数据:Metadata、"Download"-Manifest、Cart 说明: Metadata:最后一次随访的临床数据 Manifest:样本注释文件,主要用于Data Transfer Tool工具下载数据时使用 Cart:压缩包,包含所有的XML文件,也就是临床数据的压缩包文件。 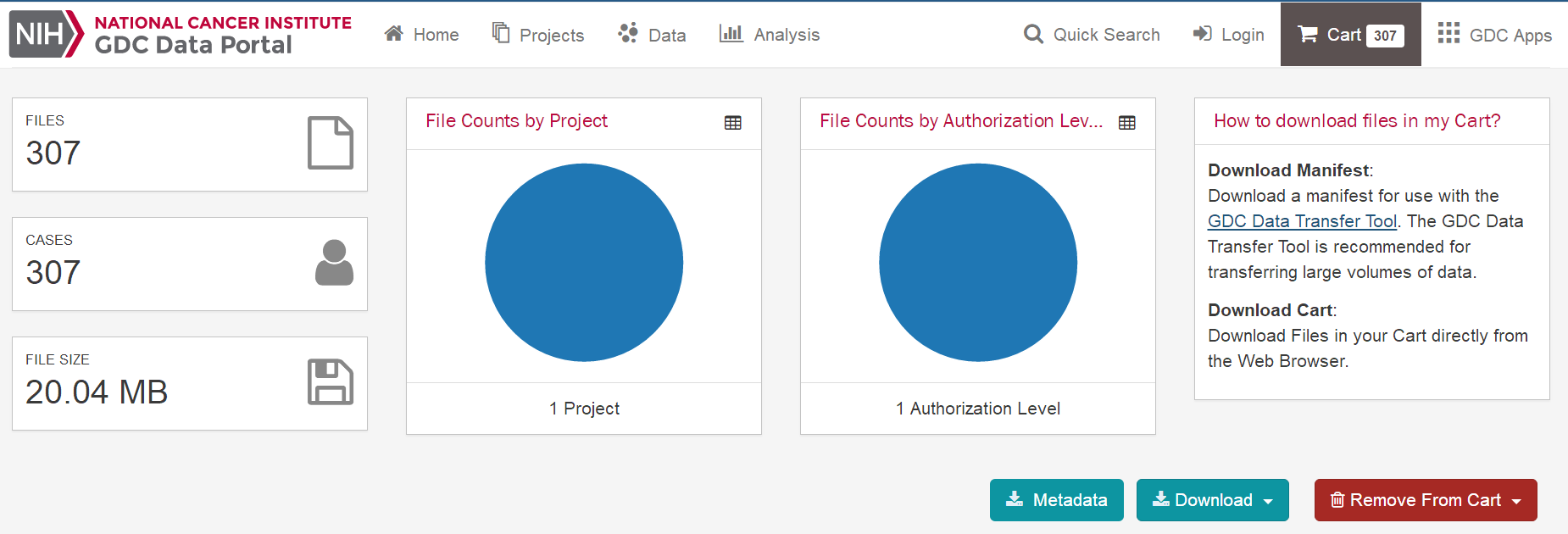 5、TCGA数据库在数据下载有规定:让Cart文件夹大于50M时(这个依据网络情况,和下载用户数目),只能通过Data Transfer Tool工具进行下载。我们这里的Cart时20.04M,一般情况可以直接下载压缩包。注:后面mRNA的内容,我们会讲解如何使用Data Transfer Tool工具下载数据。 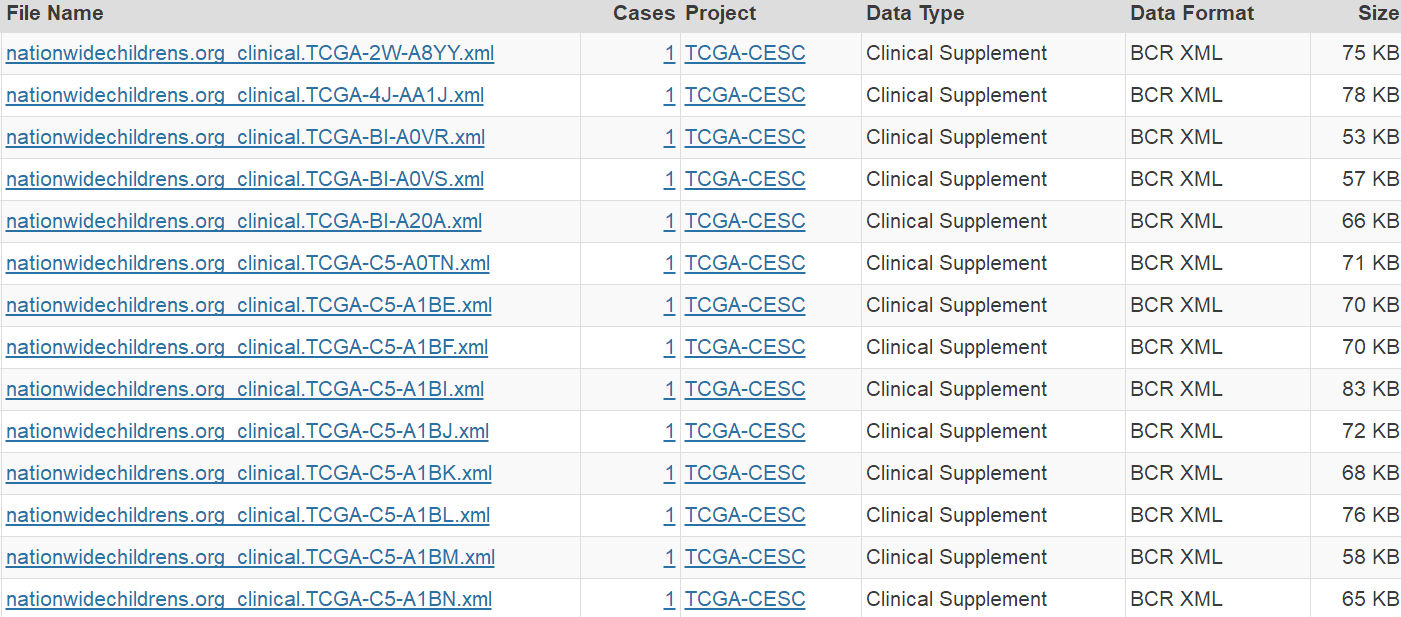 6、下载好所有需要的数据之后,我们需要用perl脚本提取文件里面的临床数据。我们首先把gdc_download_20170405_074438.tar.gz这个压缩包解压,解压得到307个文件夹,也就是一本样本一个临床数据文件夹。  7、把307个文件夹、MANIFEST.txt、get_clinical.pl脚本放在一起,我们在CMD里面输入代码"perl get_clinical.pl MANIFEST.txt",按回车,脚本文件开始运行,运行完就可以得到我们需要的clinical.txt 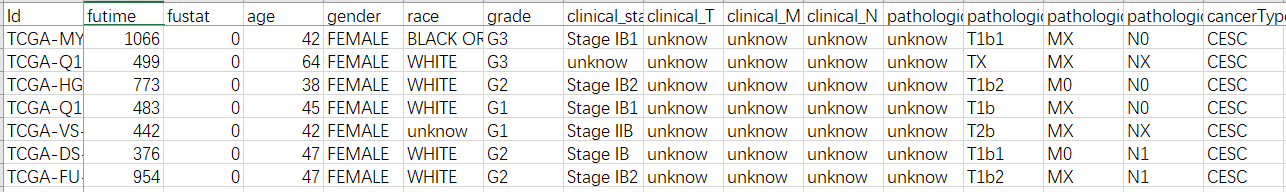
到这里,这节课需要分析的内容就讲完了,希望对大家有帮助,如果需要获得更便捷的咨询,可以关注微信公众号。 (责任编辑:伏泽 微信:18520221056) 
|
- 上一篇:用命令行下载TCGA癌症数据
- 下一篇:TCGA下载和提取miRNA表达数据