签名预测浆液性卵巢癌的总生存率(二)
时间:2020-09-23 来源:生信自学网 作者:刘鑫
微信公众号:biowolf_cn 点击:次
免疫相关基因对签名预测浆液性卵巢癌的总生存率(二)免疫细胞浸润在大体积肿瘤中为了研究不同危险人群中免疫细胞的富集情况,我们使用CIBERSORT,一种预测大块肿瘤GEPs免疫细胞浸润的机器学习方法。CIBERSORT利用支持向量回归估计了在大块肿瘤GEPs中各种免疫细胞类型的富集。对于每个样本,CIBERSORT量化了22种浸润性免疫细胞的相对丰度,其中包括T细胞、B细胞、巨噬细胞和自然杀伤细胞等。CIBERSORT使用蒙特卡罗抽样计算每个样本反褶积的p值,以提供估计的可信度。TCGA的GEPs被上传到在线分析平台CIBERSORT门户网站(http://cibersort)。stan ford.edu/),使用1,000个排列的默认签名矩阵。 基因本体(GO)与基因集富集分析(GSEA) 使用gProfiler对预后免疫签名进行GO分析(https://biit.cs.ut.ee/gprofiler/)。基因集富集分析使用具有10000个排列的生物导体包fgsea进行。取高危险组和低危险组基因表达谱的log2倍变化。比较了高危险组和低危险组的基因集。本研究涉及的生物学过程从分子签名数据库(MSigDB C2和C5数据库,版本6)(http://www.broadinstitute.org/gsea/ MSigDB /collections.jsp)下载。采用fdr调整的P<0.05或名义(NOM) P<0.05选择有统计学意义的基因集。 统计分析 所有统计检验都使用R (version 3.4.3, www.r-project.org)进行。学生t检验被用来比较组间的差异。采用Kaplan-Meier法计算累计生存时间,采用生存包(version: 2.41.3)的log-rank检验分析生存曲线差异。使用survcomp包装(版本:1.28.4)计算危险比。使用Cox比例风险回归模型进行了单变量和多变量分析。在所有检验中,pvalue小于0.05为差异有统计学意义。显著性为*P<0.05, **P<0.01, ***P<0.001。 结果预测IRGPs信号构建采用TCGA队列(n=490)基因表达数据作为发现队列,保留变异较大的基因作为候选基因,采用中位绝对偏差(MAD) >0.5进行评价。筛选后的发现数据集将使用ImmPort数据库(2018年3月18日访问)中获得的1811个独特免疫相关基因(IRGs)构建预后模型,包括17个类别,在发现集和其他平台上测量了398个IRGs。在398个IRGs中,共建立了79,003对免疫相关基因。去除变异较小(MAD =0)的IRGPs后,留下91个IRGPs作为初始候选IRGPs。然后对训练集使用Lasso Cox比例风险回归定义IRGP指数(IRGPI),并在最终的风险评分模型中选取20个IRGP。IRGPI包括40种独特的irg,其中27种是抗菌素和细胞因子相关分子(表1)。 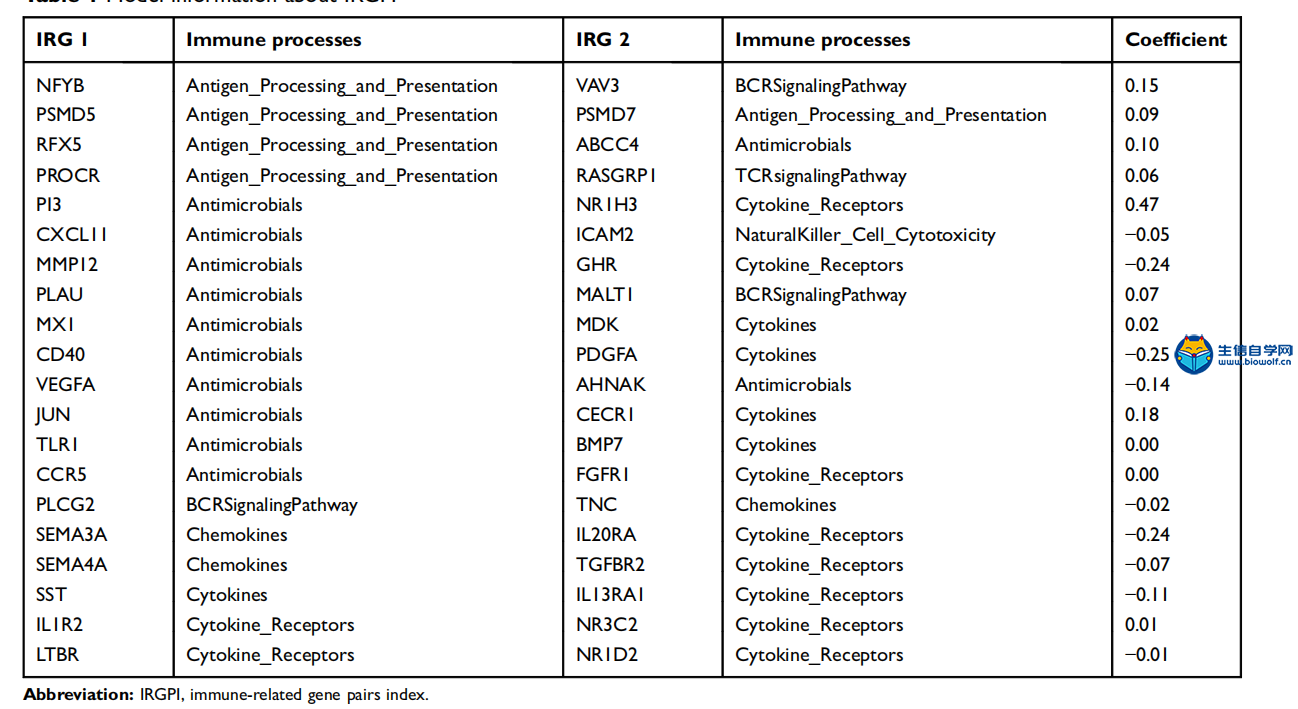 然后,我们使用IRGPI计算训练组每个患者的风险评分。根据时间依赖性ROC曲线分析,将IRGPI划分为高免疫风险组和低免疫风险组的最佳分界值为0.074(图1)。  IRGPI将训练集患者按照总生存率(OS)显著分为低免疫风险组和高风险组。我们的数据表明高风险组比低风险组表现出明显的更差的OS (P=1.44 10 14;HR =3.05[2.26, 4.10])(图2A)。  为了进一步探讨IRGPI对其他临床因素的预后作用,我们将单变量和多变量Cox比例风险回归分析应用于TCGA队列。在单变量Cox分析中,年龄和肿瘤分期等临床特征显示有预后影响。然而,在多变量Cox分析中,只有IRGPI签名仍然是一个独立的预后因素(表2)。  (责任编辑:伏泽 微信:18520221056) 
|
- 上一篇:签名预测浆液性卵巢癌的总生存率(一)
- 下一篇:签名预测浆液性卵巢癌的总生存率(三)