预后免疫基因对的筛选和模型的构建
时间:2020-09-24 来源:生信自学网 作者:刘鑫
微信公众号:biowolf_cn 点击:次
预后免疫基因对的筛选和模型的构建筛选预后免疫基因对我们将免疫基因对生生存数据合并之后,我们就可以找出哪些免疫基因对和生存是相关的,下面是对免疫基因对进行筛选后得到的表格 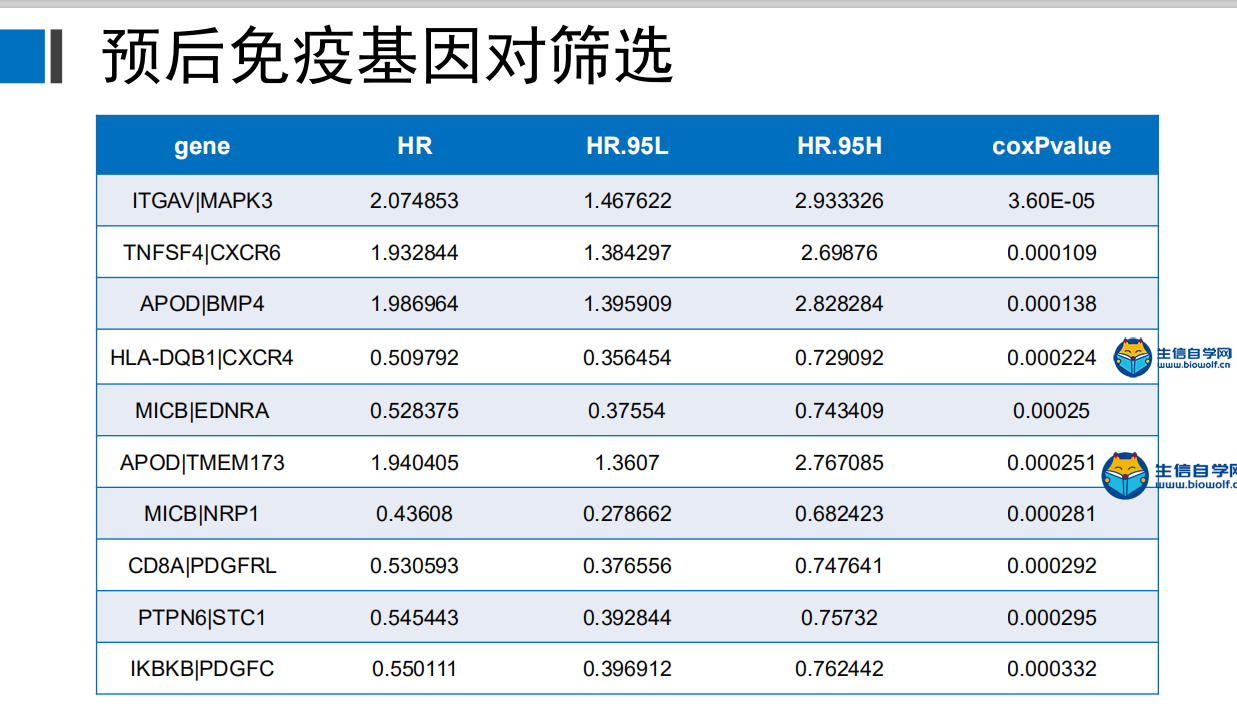 在这个表格里面它的第一列就是免疫基因对的名称,第二列是HR值,HR值如果大于1的话就说明这个基因对是高风险的免疫基因对,如果小于1的话就说明这个免疫基因对是低风险的免疫基因对。最后一列是P值,如果P值小于0.05的话就说明这个免疫基因对和生存是相关的。 下面具体操作 输入文件的话我们需要用到TCGA免疫基因对和生存时间合并后的文件。 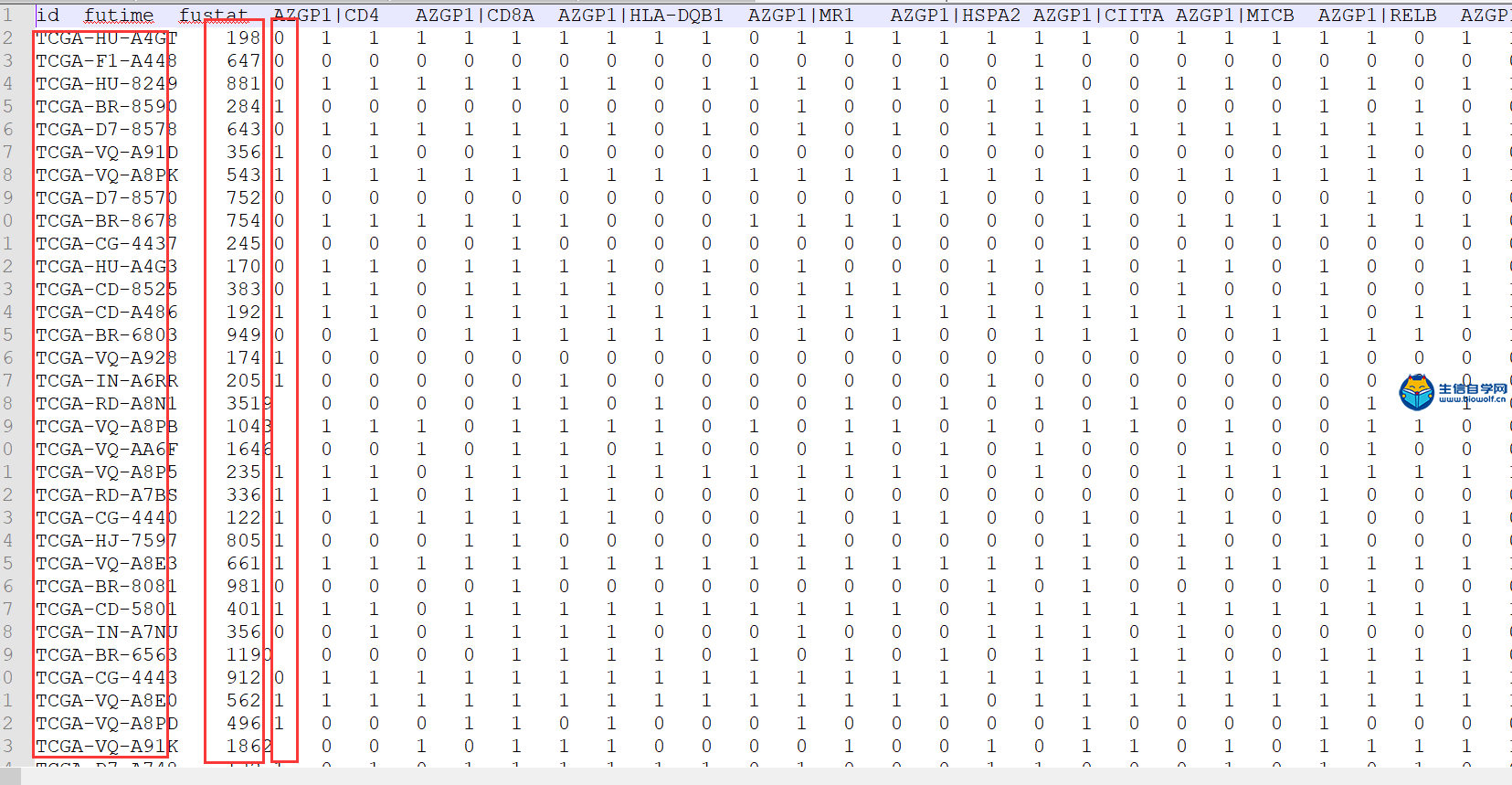 其中包含免疫基因对的名称,生存时间,生存状态和免疫基因对的状态,及表达量的比较, 然后是脚本文件,配置好脚本的工作路径后,可以在R中运行,等待运行完成后,我们就可以得到两个输出文件。一个是我们前面介绍的筛选后的表格文件,另一个是跟我们预后显著相关的免疫基因对,用于接下来构建预后相关的模型。 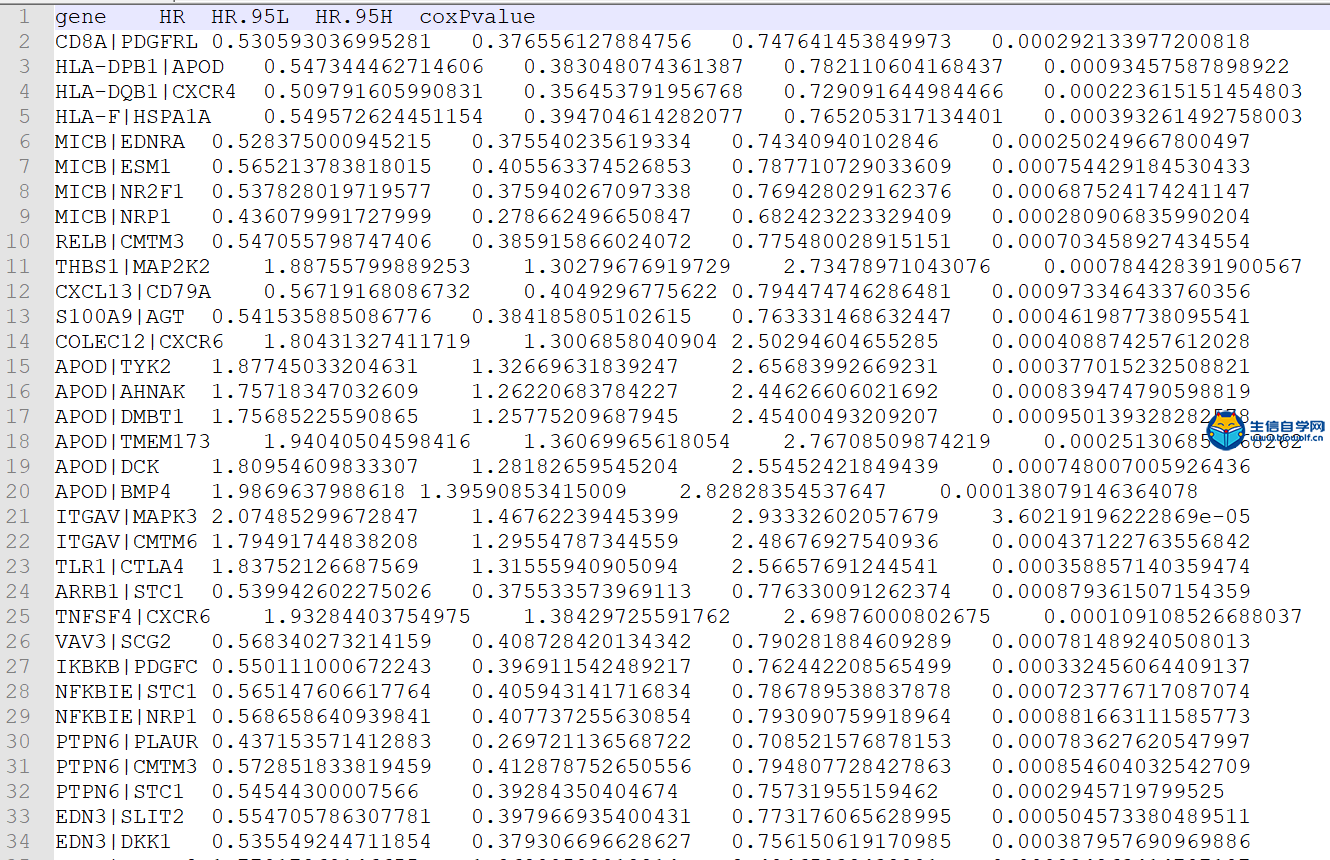 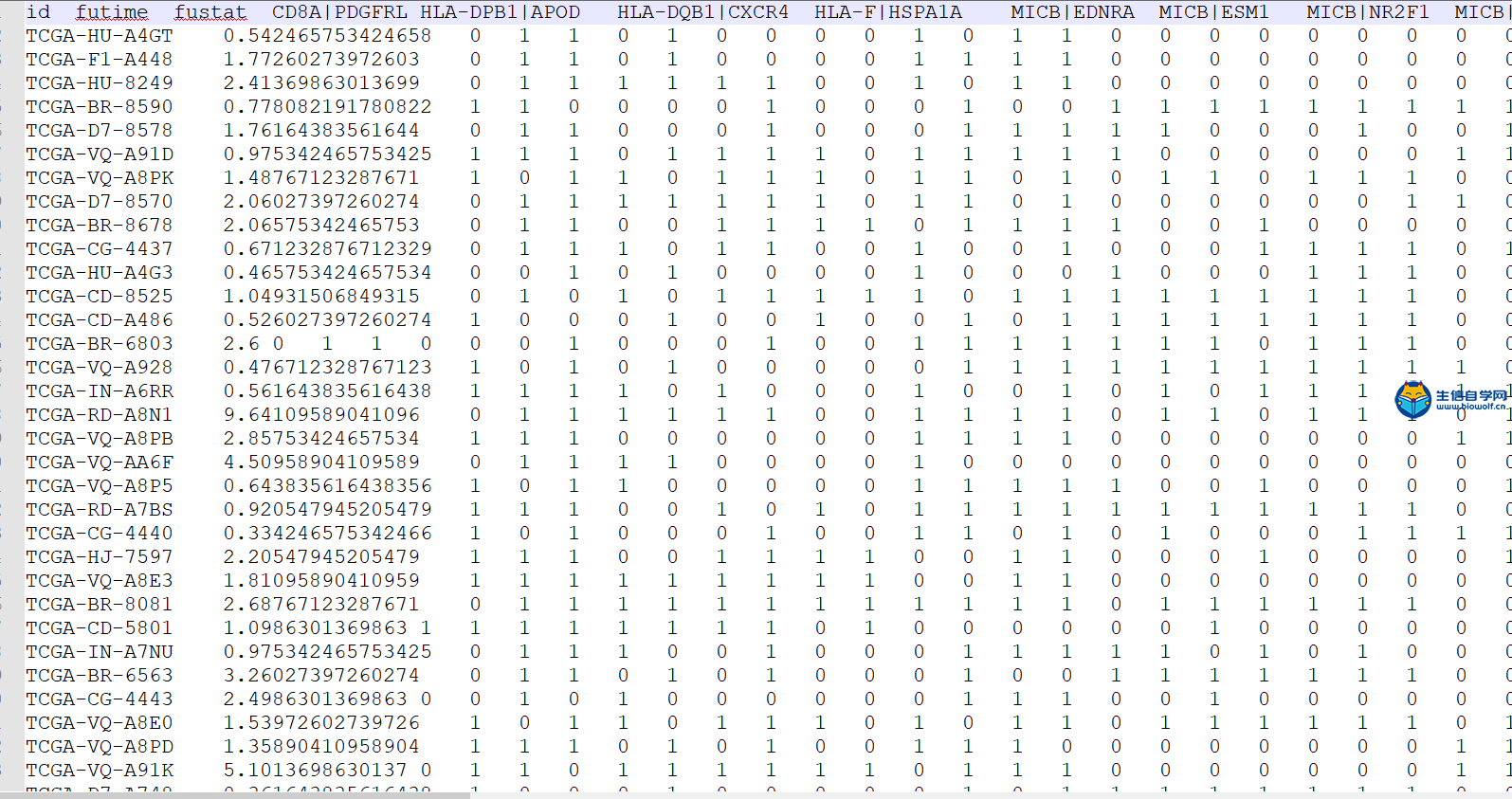 预后模型构建 得到预后相关的免疫基因对之后,下面我们就可以构建预后模型,通过下面这个过程来构建预后模型 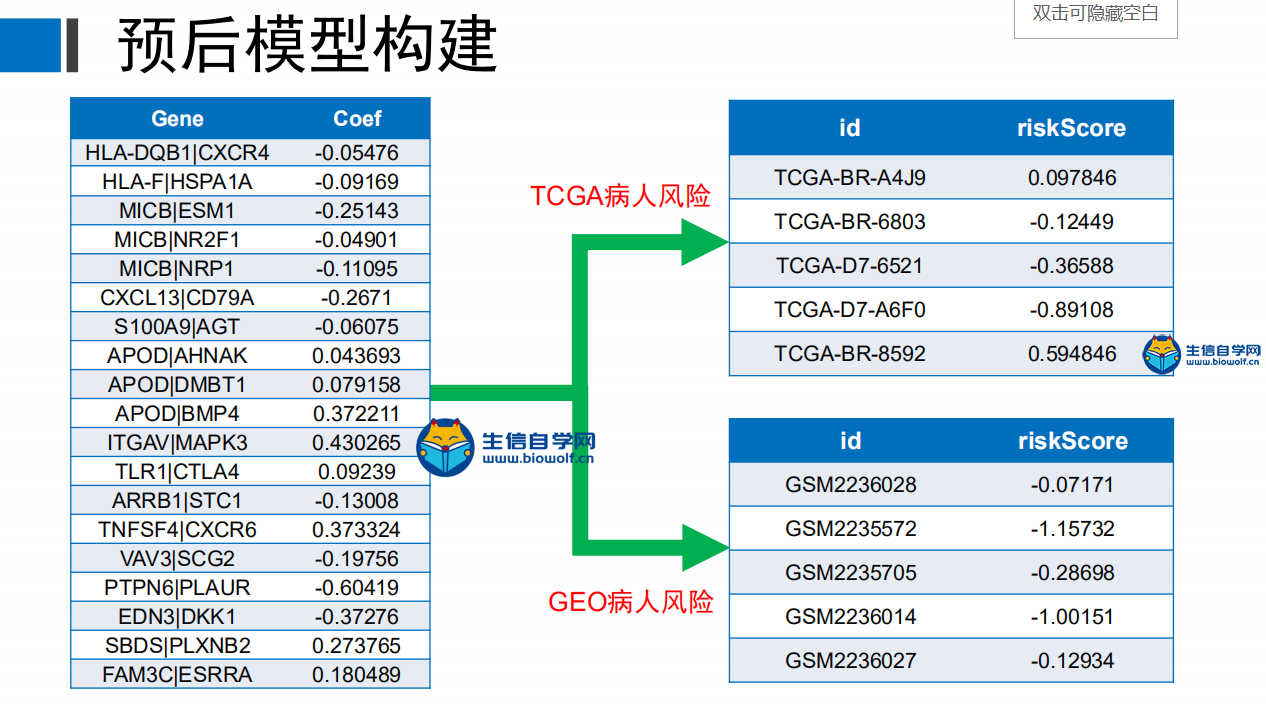 左边的两列一列是免疫基因对,一列是系数,我们用免疫基因对乘以它的系数,然后累加起来。这就是预后模型构建的公式,这样我们就可以计算每个病人的风险值,最终就会得到TCGA和GEO病人的风险值文件。 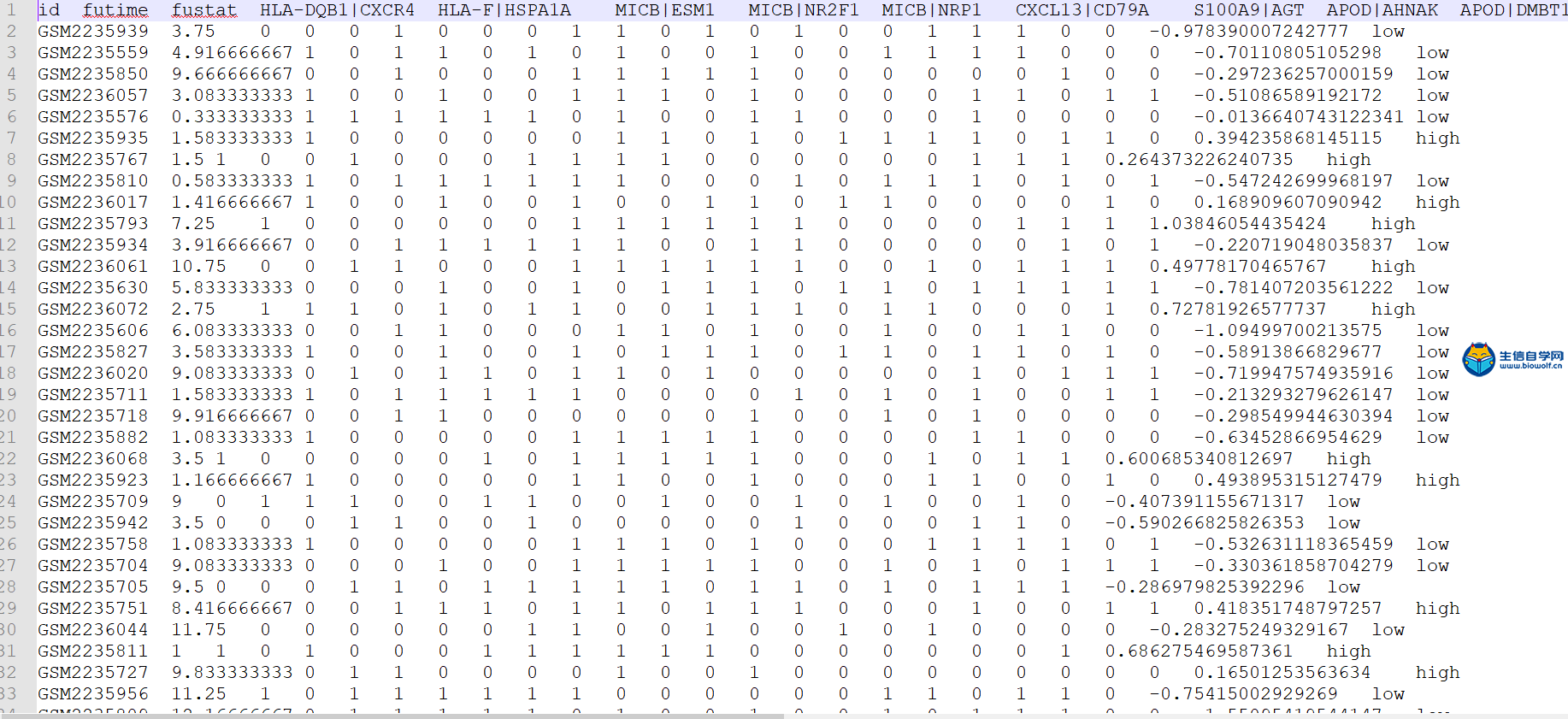 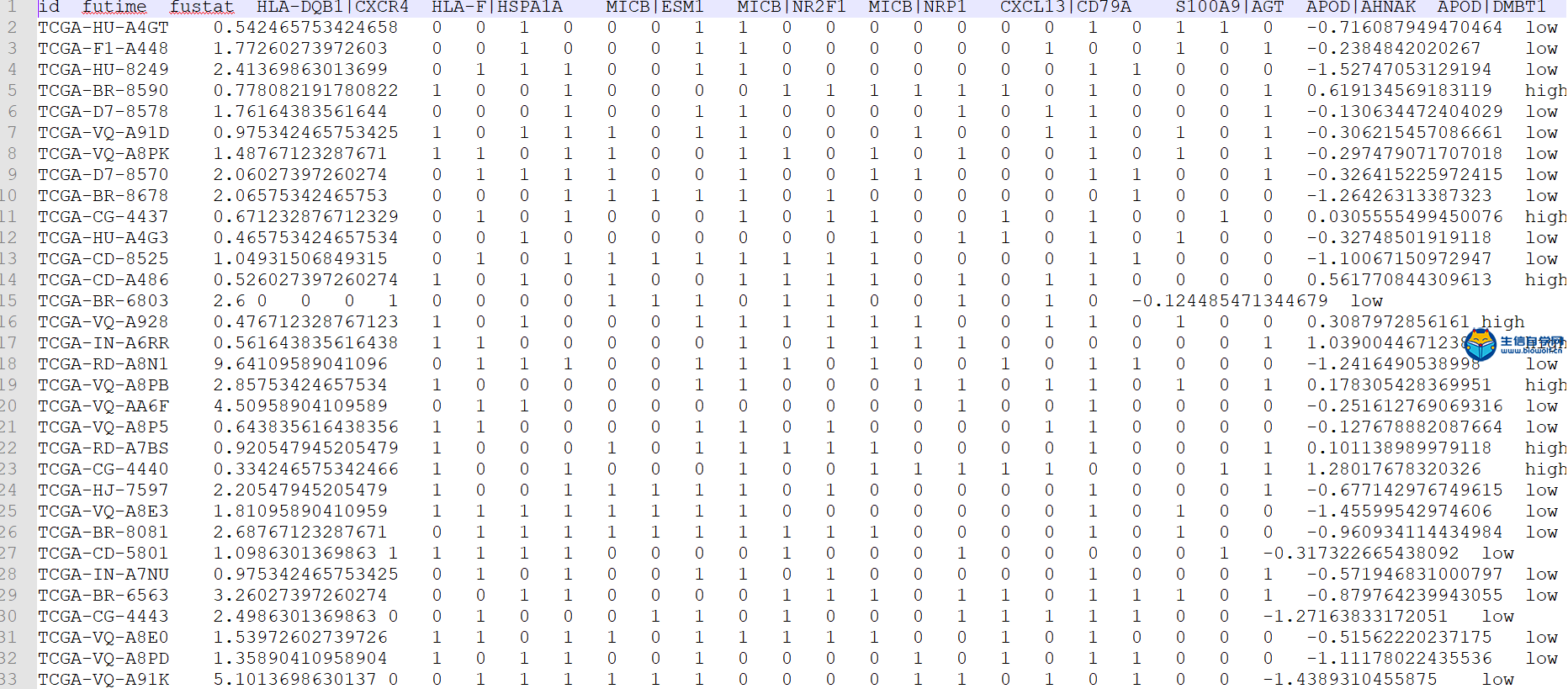 然后也可以绘制出ROC曲线,根据绘制出ROC曲线,我们可以找到最优的cutoff(下图),根据最优cutoff将病人的风险值划分为高低两组。  这个图形的横坐标是假阳性率,纵坐标是真阳性率。 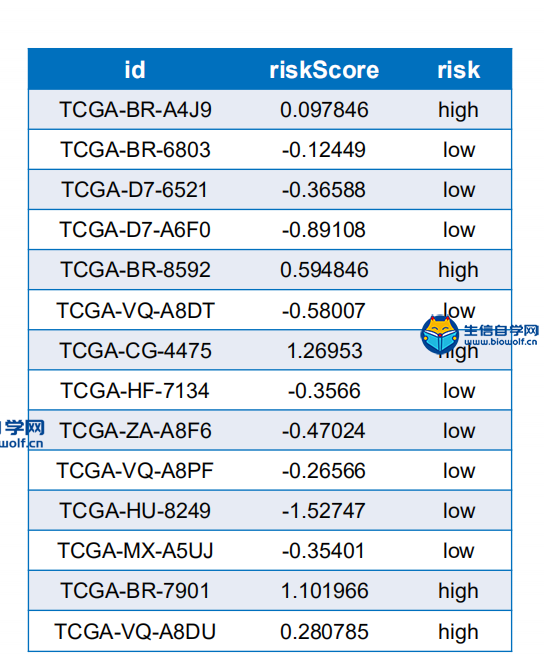 视频购买链接: 《免疫基因对文章套路视频》 精品课程推荐: 《GEO数据库miRNA芯片挖掘》 《甲基化肿瘤分型文章套路视频》 《TCGA肿瘤免疫细胞浸润模式挖掘》 (责任编辑:伏泽 微信:18520221056) 
|
- 上一篇:CEO免疫基因对和生存时间进行合并
- 下一篇:生存分析绘制生存曲线