GEO数据库免疫基因表达量的提取
时间:2020-09-22 来源:生信自学网 作者:刘鑫
微信公众号:biowolf_cn 点击:次
GEO数据库免疫基因表达量的提取在上一篇文章中我们得到了GEO数据库的表达矩阵,接下来我们要做的就是从这个GEO的表达矩阵里面,把免疫基因的表达量提取出来。提取出来GEO的免疫矩阵之后,我们还要将GEO的免疫基因和TCGA的免疫基因进行取交集的操作,因为我们后期要做模型的验证,所以我们构建模型的基因必须同时存在于两个数据库。 通过TCGA数据库提取免疫基因的表达量,我们就可以得到这样一个表格: 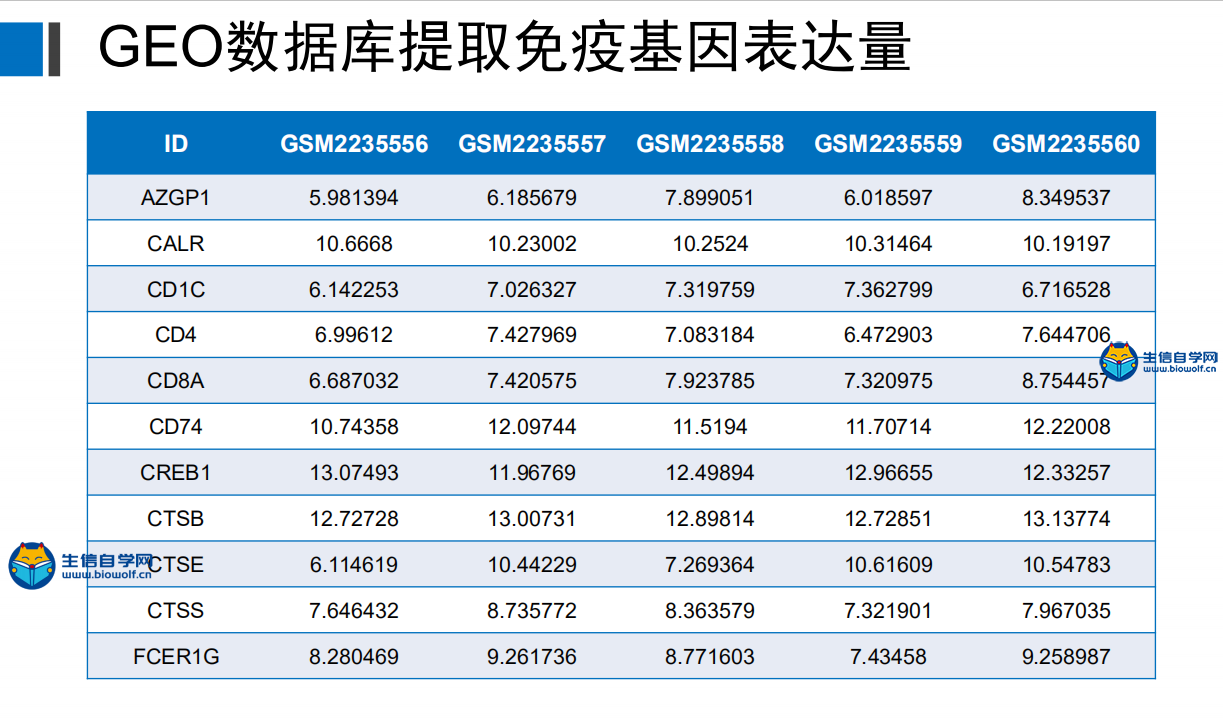 这个表格行名是基因的名称,当然这里的基因是我们的免疫基因,同时它也是和我们TCGA数据库具有交集的免疫基因。然后它的列名就是样品名称,这里面的数值就是免疫基因在我们各个样品里面的表达量。 下面我们看一下具体的操作 1、首先我们要准备我们的输入文件,我们需要用到两个输入文件,一个是GEO的表达矩阵geoMatrix.txt文件,另一个是TCGA免疫基因的表达矩阵tcgaImmuneExp.txt。我们要在geoMatrix.txt文件中把免疫相关的基因提取出来,再和tcgaImmuneExp.txt里的基因进行取交集。  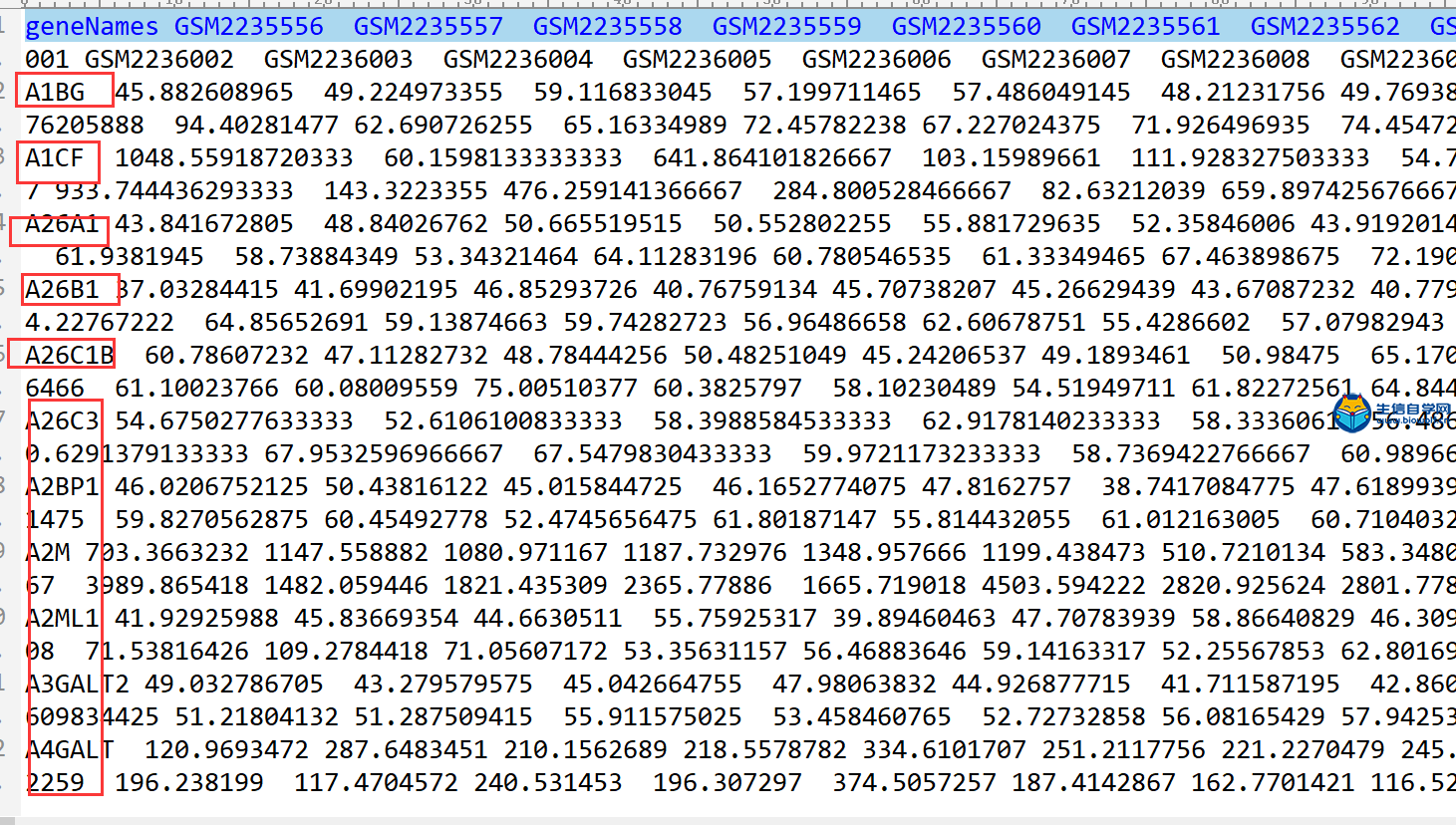  2、通过我们事先准备好的脚本文件,用R软件运行便可,脚本文件由我们生信自学网提供,大家可以购买课程获得脚本。在运行脚本之前还要导入limma包,代码如下 if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("limma") 3、运行完成后,我们会得到两个文件,这两个文件对比我们可以发现,这两个文件的基因名完全一致,这就是两个文件取交集后得到的基因。 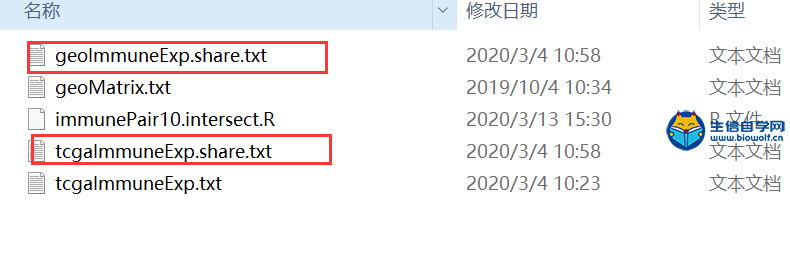 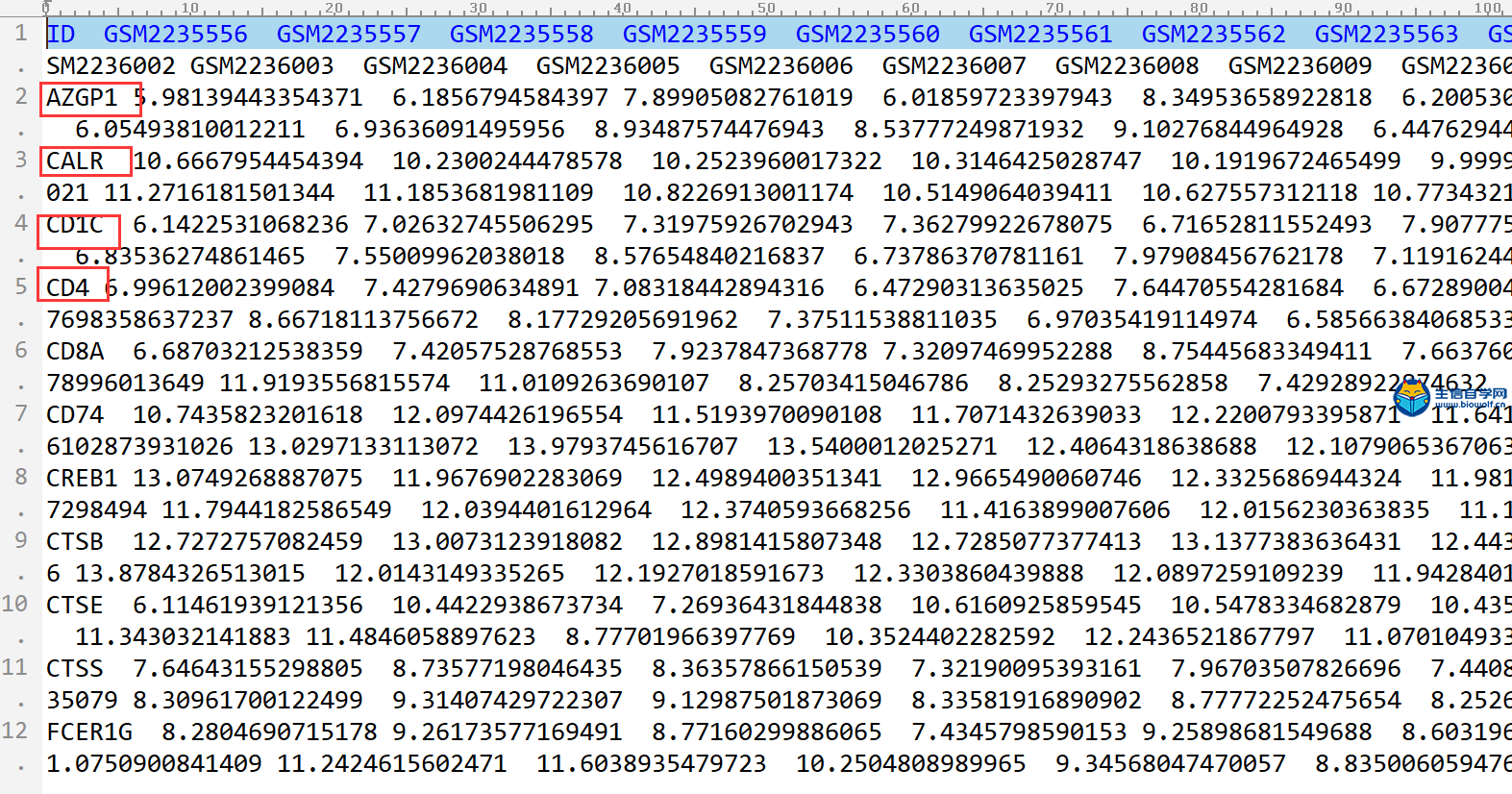  这样我们GEO提取免疫基因的表达量的操作就完成了。 视频购买链接: 《免疫基因对文章套路视频》 精品课程推荐: 《GEO数据库miRNA芯片挖掘》 《甲基化肿瘤分型文章套路视频》 《TCGA肿瘤免疫细胞浸润模式挖掘》 (责任编辑:伏泽 微信:18520221056) 
|
- 上一篇:GEO数据的注释
- 下一篇:TCGA免疫基因对和生存时间合并